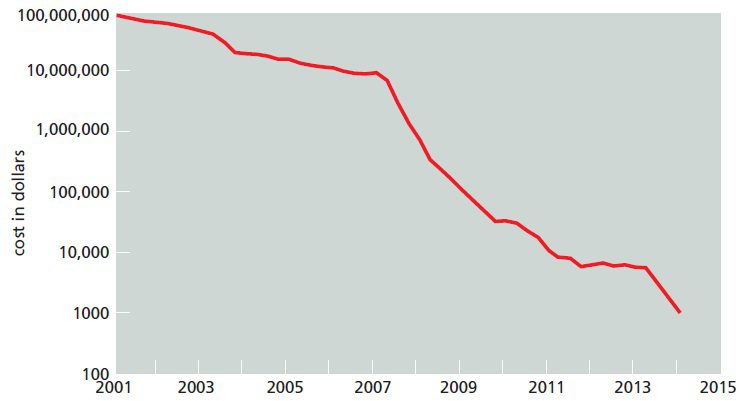
موج جدیدی از تکنولوژیها که توالی یابی نسل سوم خوانده میشوند، و بسیار سریعتر از تکنیکهای پیشین هستند، در حال توسعه اند. برخی از این تکنیکها، تمام مراحل تکثیر DNA را دور میزنند و امکان تکثیر مولکولهای DNA واحدی که تکثیر نشدهاند را فراهم مینمایند. این کار از خطاهایی که هنگام تکثیر DNA رخ میدهند، جلوگیری میکند و یا آنها را به حداقل میرساند. همچنین تکنولوژیهای مورد استفاده در این تکنیکها بسیار سادهتر از متدهایی هستند که در فرایند آنها تکثیر توالی الگو وجود دارد و با صرف هزینه کمتری نیز انجام میشوند.
مقالات مرتبط:
شرکت Helicos Biosciences نخستین تکنیک توالی یابی تک مولکولی، که به صورت تجاری موجود شد را ارائه کرد. دستگاه تولید شده توسط این کمپانی که HeliScopeTM نام داشت، در سال ۲۰۰۸ عرضه شد. از آنجایی که دستگاه توالی یاب HeliScope مولکولهای واحد را تشخیص میدهد، پس میتوان کل توالیهای مورد بررسی را روی سطح فلوسل به طور فشردهای جای داد (حدود ۱۰۰ میلیون الگوی تکمولکولی در هر سانتیمتر مربع و یا میلیاردها عدد در هر بار راهاندازی دستگاه). توالی یابی همزمان با واکنش سنتز صورت میگیرد و طی آن چندین چرخه افزودن تکنوکلئوتیدها به همراه مرحله شستوشو در بین چرخهها انجام میشود. در نتیجه میزان خوانش توالیها در هر بار ناچیز است، اما مجموع تمام توالیهای خوانده شده بسیار قابل توجه بوده و در حدود ۴۰ گیگاباز در هر بار راهاندازی به مدت ۸ روز میباشد. این میزان حتی ممکن است در آینده بیشتر نیز بشود. در این حالت، هزینه مواد اولیه نیز کمتر از توالی یابی قطعات تکثیرشده با PCR است.
تکنولوژیهای توالی یابی نسل سوم قدرتمندتری نیز در حال توسعه اند و به احتمال زیاد قرار است تحولی در فرایند توالی یابی DNA ، هم از لحاظ سرعت و هم از لحاظ هزینه، ایجاد کنند. تکنیک پیشرو در این مسیر، نوعی توالی یابی تک مولکولی DNA به نام SMRT (single-molecule real-time sequencing) است و توسط Pacific Biosciences توسعه یافته است. گفته میشود که این تکنولوژي خواهد توانست تعیین توالی را ۲۰۰۰۰ برابر سریعتر از تکنولوژیهای نسل جدید موجود در بازار انجام دهد.
تکنیک SMRT نیز توالی یابی را همزمان با سنتز DNA انجام میدهد، اما برخلاف متدهای پیشین، سنتز به صورت real-time انجام میگیرد و درنتیجه بسیار سریعتر است. SMRT میتواند در هر بار توالی یابی تکمولکول DNA، تعداد بازهای فراوانی را بخواند (۱۰ تا ۱۵ کیلوباز). تولید توالیهای بلند بسیار مهم است، چون در صورت بلندتر بودن قطعات، آسانتر میتوان توالی کل ژنوم را جمعآوری نمود. همچنین در مورد ژنومهای کوچک، کل توالی را به دست آورد، بی آن که مانند تکنولوژیهای قدیمیتر نیاز به متدهای وقتگیر و هزینهبر gap closing داشته باشیم.
به طور طبیعی در داخل سلولها، DNA پلیمرازها سنتز DNA جدید و دو برابر کردن ژنوم را در عرض چند دقیقه انجام میدهند. تکنیک SMRT به صورت real-time، آنزیمهای DNA پلیمرازی که سنتز مولکولهای DNA جدید از مولکول الگوی واحد که تکرشتهای است را انجام میدهند، تحت نظر میگیرد. در این حالت تشخیص نوکلئوتیدهای متصل شده به هر جایگاه با استفاده از نوکلئوتیدهای علامتگذاری شده با یکی از چهار فلوروفور متفاوت و طبق واکنش اختصاصیت باز انجام میشود.
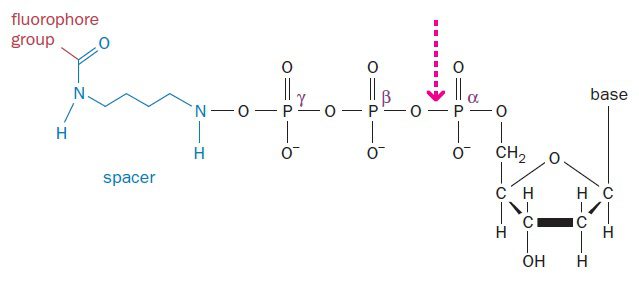
SMRT از دو نوآوری کلیدی بهره میگیرد. نخست اینکه فلوروفورها به طرز غیرمعمولی به dNTP ها لیبل شدهاند. در حالت عادی، فلوروفورها به نحوی به بازها متصل میشوند که با اتصال باز به ساختار DNA، جزئی دائمی از رشته DNA میگردند. در این شرایط، پس از افزوده شدن چندین نوکلئوتید به DNA، توده فلوروفورها به علت فضایی که اشغال میکند از سنتز DNA توسط آنزیم پلیمراز جلوگیری مینماید. به همین دلیل در بسیاری از متدهای توالی یابی که همزمان با سنتز صورت میگیرند، در هر بار، سنتز تنها یک نوکلئوتید انجام میشود و پس از آن واکنش خاتمه مییابد. در این حالت به مقادیر فراوانی از مواد اولیه نیاز خواهیم داشت و processivity آنزیم نیز بسیار محدود است.
در مقابل، در تکنیک SMRT، فلوروفورها به جای خود باز، به گروه فسفات γ که خارجیترین گروه در بخش تریفسفات نوکلئوتیدها است، متصل میشوند. هنگامی که چنین نوکلئوتیدی با باز مکملش در توالی الگو جفت میشود، میتوان سیگنال فلورسنت را پیش از برش گروه تریفسفات توسط پلیمراز، ضبط نمود؛ یعنی در لحظهای که dNTP هنوز متصل به جایگاه فعال آنزیم است. نوکلئوتیدهایی که مدت زمان طولانیتری به آنزیم متصل میمانند، مکمل هستند. سپس dNMP بدون لیبل، در اثر برش تولید شده و به ساختار ژنوم میپیوندد. در نتیجه پلیمراز میتواند بدون وقوع مشکلات مربوط به شکل فضایی به افزودن نوکلئوتیدها ادامه دهد و باعث تولید خوانشهای بلندی از توالی شود.
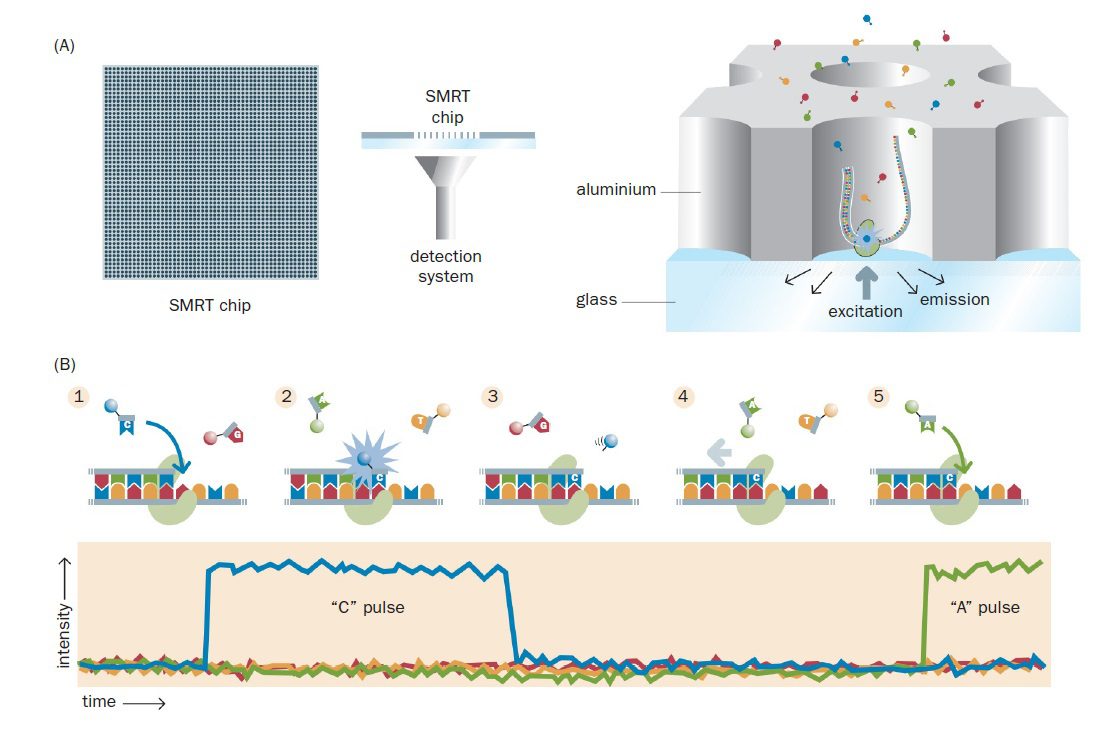
نوآوری مهم دوم، چیپ SMRT است و صفحه فلزی بسیار نازکی با پایه شیشهای میباشد. صفحه فلزی حدود ۱۰۰ نانومتری، حاوی یک میلیون حفره sub-wavelength به نام ZMW نانوفتونیک (nanophotonic zero-mode waveguide) است که قطری در حد یک دهم نانومتر دارند و هر کدام قادر به نگهداری یک مولکول DNA الگو میباشند. البته طبق توزیع Poisson، از یک میلیون حفره، تنها حدود ۳۳۰۰۰۰ عدد از آنها دارای توالی میگردند.
هر ZMW یک محفظه مصورسازی نانوفتونیک (nanophotonic visualization chamber) را تشکیل میدهد و DNA پلیمراز متصل به تکرشته الگو به کف پایه شیشهای متصل است. میتوان از روی پایه شیشهای ZMW، به طور مستقیم DNA پلیمراز را در حال انجام توالی یابی به صورت همزمان با همانندسازی، روی تکمولکول DNA مشاهده کرد. امواج نور جمعآوری شده از رشتههای در حال همانندسازی بررسی و به طور موازی آنالیز میشوند. با انجام یک سری اصلاحات، این تکنیک احتمالا خواهد توانست کل ژنوم انسان را در عرض یک ساعت و با صرف هزینهای در حدود صد دلار توالی یابی کند.
یکی دیگر از پیشرفتهای مهم در تکنیکهای توالی یابی نسل سوم ، توالی یابی nanopore است. موادی مانند سیلیکون میتوانند به نحوی شکل داده شوند که دارای حفرات بسیار ریزی گردند. در این حالت، DNA از چنین لوله باریکی عبور داده میشود (مشابه نخ کردن سوزن)؛ به نحوی که توالیهای مولکول تک رشتهای DNA به ترتیب از درون لوله عبور کنند. همزمان با این کار، مولکول DNA، بسته به ماهیت هر باز، تولید جریان الکتریکی متفاوتی میکند. الگوی جریانات میتواند برای استخراج توالی استفاده شود. اساس تولید جریان الکتریکی، مسدود شدن نانوپور در درجات و خصوصیات متفاوت توسط بازهای مختلف است.
با معرفی چنین تکنیکهای توالی یابی حساس و ارزانی، دانشمندان میتوانند شروع به توالی یابی مجدد ژنومها بکنند تا به دقت بالاتری دست یابند. به عنوان مثال، با استفاده از SMRT، توالی ژنوم E. coli با دقتی در حدود ۹۹.۹۹۹۹ درصد تعیین شده است. توالی یابی ژنوم انسان به این طریق هنوز ممکن نیست؛ اما زمانی که این امکان فراهم شود، احتمالا بتوانیم کل ژنوم را در حدود یک ساعت تعیین توالی کنیم و تمام این دستاوردها تنها پس از گذشت کمی بیش از یک دهه از اتمام پروژه ژنوم انسانی عملی گشته است!