چگونه میتوان یک سوزن را داخل یک انبار کاه پیدا کرد؟! با وجود میلیون ها قطعه هماندازه و هم شکل کاه، راه حل این معمای قدیمی چیست؟ آیا میتوانیم سوزن را سمت خود بیاوریم، بی آن که مجبور باشیم لابهلای ذرات کاه را جستوجو کنیم؟ بله، یک آهنربای بسیار قوی قطعا قادر به این کار هست.
با اینکه DNA ابدا شبیه یک انبار کاه نیست، اما یافتن قطعهای مشخص در یک نمونه مخلوط از آن به سختی یافتن یک سوزن در انبار کاه است. در چنین مخلوطی، تمام DNAها تقریبا اندازه، ترکیب شیمیایی و شکل مشابهی دارند، در نتیجه یافتن قطعه کوچک دلخواهمان دشوار خواهد بود. خوشبختانه دانشمندان از ویژگیهای بیوشیمیایی DNA استفاده کرده و تکنیکی به نام هیبریداسیون DNA یا DNA hybridization را ایجاد کردهاند که میتواند به این منظور استفاده شود. در هیبریداسیون DNA، توالیهای شناختهشده نوکلئیکاسیدها (پروبها) به منظور تعیین توالیهای مرتبط موجود در نمونه به کار میروند.
در سطوح ابتدایی تحقیقات، هیبریداسیون نوکلئیکاسیدها به منظور ردیابی رونوشتهای RNA و بررسی میزان بیان ژن استفاده میشود. همچنین این تکنیکها به منظور تعیین ارتباط میان توالیهای DNA ای که دارای منابع مختلف هستند، به کار میروند. به عنوان مثال، یک توالی DNA آغازگر (starting) میتواند توالیهای مشابه را در ارگانیسمهای مختلف، افراد متفاوت موجود در یک گونه و یا حتی توالی های مشابه موجود در همان ژنومی که DNA آغازگر از آن منشا گرفته است را شناسایی کند. از دیگر کاربردهای تکنیکهای هیبریداسیون نوکلئیکاسیدها، تعیین اللهای عامل بیماری و رونوشتهای غیرطبیعی مرتبط با بیماری است.
اساس هیبریداسیون DNA
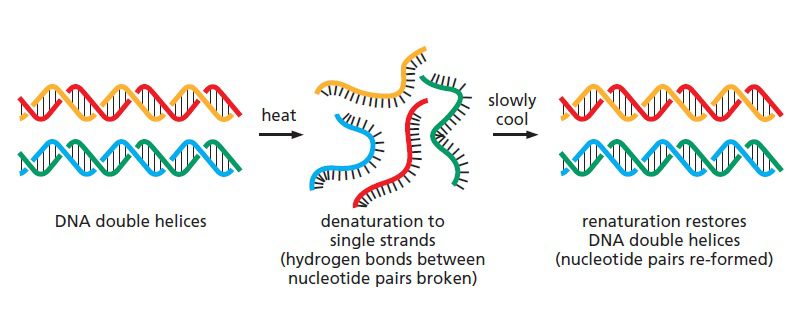
در حالت عادی،DNA متشکل از دو رشته است که با پیوند هیدروژنی بین بازهای مکمل به همدیگر متصل هستند. این پیوندها میتوانند به راحتی گسسته شوند و دو رشته جدا از هم DNA حاصل شود. این عمل دناتوراسیون DNA نام دارد وآسانترین راه برای شکستن این پیوندها، حرارت دادن DNA تا دمای حدودا ۹۰ درجه سانتیگراد است. اگر دما از این میزان به آرامی پایین آورده شود، رشتههای مکمل دوباره به همدیگر میپیوندند تا مارپیچ دو رشتهای DNA دوباره حاصل شود.
این پدیده، یعنی تشکیل پیوندهای هیدروژنی بین جفتبازهای مکمل اسید نوکلئیک، هیبریداسیون (hybridization) نام دارد و اساس آن، اختصاصیت تشکیل جفتباز است. این جفتشدن علاوه بر دو رشته DNA، میتواند بین دو رشته RNA و یک DNA و یک RNA نیز انجام شود. پس تکنیکهای هیبریداسیون DNA، از توانایی نوکلئیکاسیدهای تکرشتهای در تشکیل جفتباز با توالیهای مکمل و یا نیمه مکمل استفاده میکند.
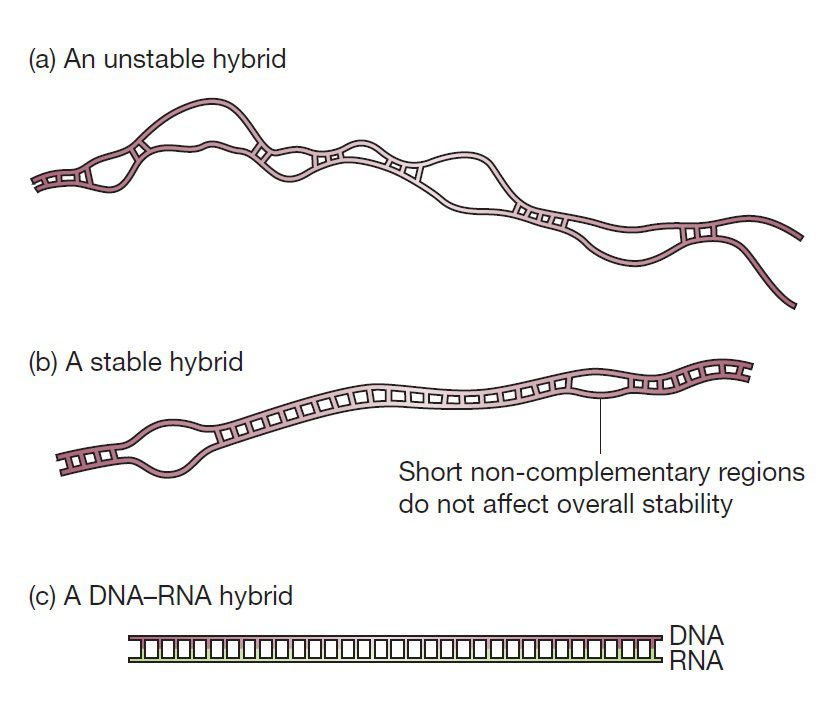
ذکر این نکته ضروری است که هر دو مولکول تکرشتهای نوکلئیک اسید، توانایی تشکیل جفتباز با یکدیگر را دارند. در مورد بسیاری از این رشتههای متصلشده، ساختار هیبریدی تشکیلشده ناپایدار است؛ چون تعداد پیوندهای درون رشتهای که به درستی جفت شدهاند، بسیار اندک است. درحالیکه اگر رشتههای پلینوکلئوتیدی مکمل هم باشند، تشکیل جفتبازها به صورت گستردهای رخ داده و یک مولکول دورشتهای پایدار تشکیل میشود.
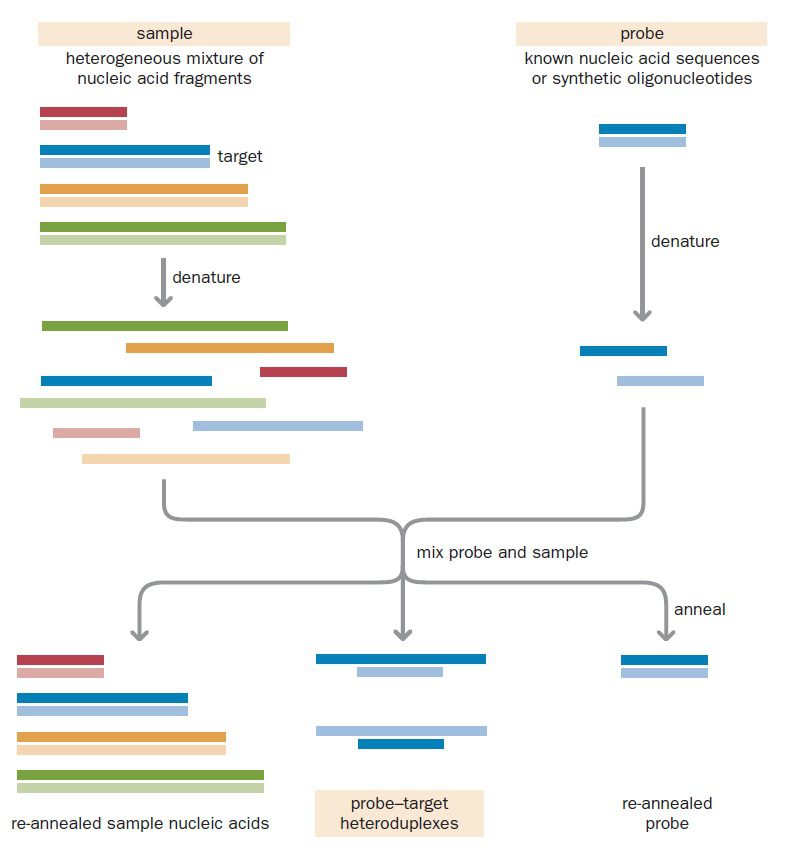
به منظور رسیدن به این هدف، نوکلئیکاسیدهای موجود در هر دو نمونه پروب و تست، ابتدا به صورت تکرشتهای در میآیند. نمونه میتواند حاوی RNA و یا DNA باشد که در هر دو حالت به دناتوراسیون نیازمندیم. از جمله روشهای انجام این کار، علاوه بر حرارت دادن مولکولهای DNA، استفاده از مواد قلیایی است. همچنین در صورت نیاز میتوان مولکولها را تحت تاثیر مولکولهای با قطبیت بالا مانند فرمآمید و اوره نیز قرار داد تا بتوانند مدت زمان بیشتری تکرشتهای بمانند.
نمونههای تست و پروب دناتورهشده، سپس مخلوط میشوند، تا اینکه در اثر اتصال توالیهای هدف و پروبها، هترودوپلکس تشکیل شود. هترودوپلکس، ساختاری دورشتهای از اسیدهای نوکلئیک است که از تشکیل جفتباز میان دو مولکول تکرشتهای حاصل شده است. این دو مولکول از اللهای یکسانی منشا نگرفتهاند. تشخیص هترودوپلکسها در اثر هیبریداسیون گروهی از نوکلئیکاسیدهای لیبلشده که محلول در آب هستند، با گروهی از نوکلئیکاسیدهای لیبلنشده که به یک پایه جامد متصل شدهاند، انجام میگیرد.
احتمال شناسایی هترودوپلکسها در محیط محلول بسیار پایین است و در صورت اتصال آنها به یک پایه جامد مثل یک غشای پلاستیکی و یا اسلاید شیشهای، میتوان به میزان زیادی این عمل را تسهیل نمود. معمولا نوکلئیکاسیدهای موجود در نمونه مورد آزمایش به پایه متصل و پروبها به صورت محلول و لیبلشده هستند.
لیبل کردن نوکلئیکاسیدهای محلول، با الحاق نوکلئوتیدهای حاوی رادیوایزوتوپها یا گروههای شیمیایی تغییریافته، به طوری که بتوانند توسط فرایندی مناسب تشخیص داده شوند، انجام میشود. سپس این محلول روی پایه جامد افزوده میشود؛ به نحوی که دو گروه از مولکولها بتوانند با یکدیگر فعل و انفعال داشته باشند. درنتیجه مولکولهای DNA موجود در نمونه آزمایش، به پروبهای لیبلشدهای که دارای توالی مکمل یا نیمه مکمل هستند، متصل خواهند شد. البته اتصالات غیراختصاصی نیز میتوانند میان پروب و پایه و یا مولکولهای غیرهدف رخ دهند. پس از شستوشوی گسترده و درنتیجه زدوده شدن نوکلئیک اسیدهای هیبریدنشده، تمام لیبلهایی که روی پایه جامد باقی مانده اند، مربوط به هترودوپلکس تشکیلشده میان پروب و توالی هدف هستند.
پایداری هترودوپلکسها به اندازه نواحی جفتشده، دما و غلظت یونهای محیط بستگی دارد. هر چه طول ناحیه جفتشده بیشتر باشد، پیوندهای هیدروژنی نیز بیشتر هستند و درنتیجه به انرژی بیشتری برای شکستن پیوندها نیاز خواهیم داشت. البته تاثیر طول در اندازههای کوچکتر نواحی جفتشده بیشتر حائز اهمیت است. تعداد نواحی جفتشده به صورت نادرست نیز پایداری هترودوپلکس را تحت تاثیر قرار میدهند. ترکیب بازها و ترکیب شیمیایی محیط حاوی مولکولهای DNA نیز مهم است. هر چه تعداد جفتبازهای GC بیشتر باشند، شکستن پیوندها سختتر و پایداری هترودوپلکس بیشتر خواهد بود. یونهای مثبت تک ظرفیتی مانند Na+ پایداری پیوندهای هیدروژنی در مولکولهای دورشتهای را افزایش میدهند و مولکولهای به شدت قطبی در ساختار این پیوندها اختلال ایجاد کرده و به عنوان ماده دناتورهکننده عمل میکنند. افزایش دما نیز میتواند پیوندهای هیدروژنی را سست کند.
در فرایند هیبریداسیون DNA، شرایط در ابتدا به نحوی تنظیم میشوند که بیشترین تشکیل هترودوپلکس صورت بگیرد، حتی اگر این هدف به قیمت وجود هیبریداسیونهای غیراختصاصی تمام شود. به عنوان مثال دمای هیبریداسیون، ۲۵ درجه سانتیگراد پایینتر از دمای ذوب تنظیم میشود؛ درنتیجه مولکولهای پروب میتوانند حتی با نوکلئیک اسیدهایی که توالی غیرمرتبطتری دارند، جفت شوند. سپس شستوشوهای پیدرپی در شرایطی انجام میشوند که رفتهرفته در تحمل جفت شدنهای نادرست ناموفقتر میباشند. به عنوان مثال دما پیوسته افزایش و یا غلظت NaCl در بافر پیوسته کاهش مییابد. در نهایت اختصاصیترین هترودوپلکسها روی پایه باقی خواهند ماند.
کاربرد هیبریداسیون DNA در تحقیقات پزشکی
۱.تشخیص میکروارگانیسمهای بیماریزا:
کاربرد پروبهای نوکلئیکاسیدی خصوصا در میکروبشناسی بسیار مهم است و میتوان از آنها در شناسایی میکروارگانیسمهای غیرقابل کشت و یا در تشخیص سریع پاتوژنها استفاده کرد. با توسعه روشهای هیبریداسیون DNA-DNA و DNA-RNA و نیز متدهای DNA نوترکیب، استخراج توالیهای اختصاصی هر گونه ممکن شده است. امروزه پروبهای الیگونوکلئوتیدی که مکمل هر یک از توالیهای موجود در زیرواحدهای بزرگ و کوچک ریبوزوم و یا نواحی internal transcribed spacer (توالی DNA مابین ژن rRNA زیرواحد بزرگ و کوچک) باشند، برای انواع زیادی از میکروارگانیسمها تولید میشوند. تشخیص وجود یک توالی نوکلئیکاسیدی یک میکروارگانیسم خاص، نشاندهنده وجود آن میکروارگانیسم در نمونه است و احتمالا میتواند وجود عفونت را تایید کند. از جمله میکروارگانیسمهایی که این روش قادر به شناساییشان است، عبارتند از: Actinomyces ،Bacteriodes ،Borrelia ،Campylobacter ،Clostridium ،Candida ،Haemophilus ،Helicobacter ،Lactococcus، Mycoplasma ،Neisseria ،Proteus ،Vibrio ،Rickettsia ، Plasmodium ،Streptococcus ،Pneumocystis ،Trichomonas ،Desulfovibrio و Streptomyces .
۲. تشخیص تغییرات توالیهای نوکلئیک اسید:
هر گونه تغییر در توالی DNA جهش نام دارد و از انواع آن، حذف، درج و یا جانشینی است. تغییر یافتن ژنهای مشخصی با بیماریهای ارثی مانند فیبروز سیستیک (cystic fibrosis)، دیستروفی عضلانی، فنیل کتونوریا، تفاوتهای ژنهای تولیدکننده آپولیپوپروتئینها (apolipoprotein variants) و آنمی داسی شکل مرتبط است. پروبهای نوکلئیک اسیدی میتوانند به خوبی این جهشها را شناسایی کنند. در برخی از بیماریهای ارثی، بیش از یک جهش عامل بیماری است. در اینگونه موارد، باید پروبی به کار رود که اختصاصیت کمتری داشته باشد (low stringency)، به این معنی که بتواند با انواعی از توالیها هیبرید شود. همچنین میتوان از چندین نوع پروب استفاده کرد تا مطمئن شویم که همه توالیهای هدف به پروبها متصل شدهاند.
۳.تشخیص توالیهای تکراری پشت سرهم (tandem repeat sequences):
توالیهای tandem repeat معمولا طولی در حدود ۵۰-۳۰ جفتباز دارند. اندازه و توزیع آنها در هر فرد اختصاصی است. این توالیها میتوانند با استفاده از پروبهای نوکلئیک اسیدی و PCR شناسایی شوند و اساس تکنیک انگشتنگاری DNA را تشکیل میدهند. این تکنیک در پزشکی قانونی برای تایید هویت مظنونین و از روی نمونههای به جای مانده در صحنه جرم مانند مایعات بدن، پوست و مو، به کار میرود. همچنین از کاربردهای دیگر آن، تستهای تشخیص هویت و tissue typing (تست تعیین سازگاری بافتی گیرنده و دهنده در پیوند عضو) میباشد.
مقالات مرتبط:
- پروب های نوکلئیک اسیدی
- تکنیک Southern Blotting
- تکنیک Northern Blotting
- تکنیک FISH
- تکنیک DNA microarray

عالیه.فقط اگر منابع هم میذاشتین بهتر بود.
اگر امکانش هست منابعش برای من ارسال بفرمایید.
خیلی عالی نوشتید خسته نباشید از مقاله لذت بردم