معرفی توالی یابی نسل جدید (Next Generation Sequencing= NGS) در سال ۲۰۰۷ تحولی در دنیای ژنومیکس پدید آورد، روشی کاملا جدید برای تعیین توالی DNA معرفی نمود و هزینه و زمان موردنیاز برای توالی یابی ژنوم را به شدت کاهش داد. مثالی که برای اهمیت این روش میتوان عنوان کرد آن است که پروژه ژنوم انسان ۱۰ سال طول کشید و بیش از ۳ میلیارد دلار بودجه مصرف کرد؛ این در حالی است که امروزه توالی یابی نسل جدید میتواند در عرض یک روز کل ژنوم انسان را با صرف هزینهای در حدود ۵۰۰۰ دلار توالی یابی کند. یکی از علل این موضوع آمادهسازی سریعتر نمونه نسبت به تکنیکهای پیشین است؛ به عنوان مثال دیگر نیازی به تولید کتابخانههای DNA در باکتریها نیست.علاوه بر صرفهجویی در هزینه و زمان، پس از کشف این تکنیکها تعداد ژنومهای توالی یابی شده نیز افزایش قابل توجهی داشته است. دو متد اصلی برای این نوع از توالی یابی وجود دارد: توالی یابی ۴۵۴ و Illumina.
مقاله مرتبط: توالی یابی چیست؟
این تکنیکها نیازی به الکتروفورز ندارند و ثبت توالی DNA را همزمان با سنتز آن از DNA الگوی تکرشتهای انجام میدهند. به عبارت دیگر در این تکنیکها، اتصال هر کدام از انواع نوکلئوتیدها به زنجیره DNA در حال رشد بررسی میشود. توالی یابیهای نسل جدید از پلتفرمهایی استفاده میکنند که میتوانند میلیونها قطعه DNA را در موقعیتهایی جداگانه ثابت و سپس آنالیز کنند و درنتیجه چندین ژنوم میتوانند به طور موازی در عرض کمتر از یک هفته توالی یابی شوند. همچنین این تکنیکها بسیار انعطافپذیر هستند و میتوانند برای تمام انواع و اندازههای مختلف ژنوم، از ویروس گرفته تا انسان به کار روند.
طبق موارد گفته شده، این تکنیکها این امکان را برای پژوهشگران پدید میآورد که ژنوم هزاران انسان را بررسی کنند، تفاوتهای موجود در توالی نوکلئوتیدی افراد در سراسر جهان را بیابند و پرده از جهشهایی که ریسک ابتلا به بیماریهای مختلف از سرطان تا اوتیسم را افزایش میدهند، بردارند. تکنیکهای توالی یابی نسل جدید همچنین امکان تعیین توالی ژنومی گونههای منقرض شده مانند نئاندرتالها و ماموتها را فراهم میکند. همچنین با توالییابی گونههای نزدیک به هم، درک اساس مولکولی رخدادهای کلیدی تکاملی در درخت زندگی، مانند چندسلولی شدن، بینایی و زبان آسانتر شده است. افزایش سرعت توالییابی تاثیراتی شگرف بر شاخههای مختلف بیولوژي و پزشکی گذاشته است؛ تا حدی که تصور وضعیت این علوم بدون آنها ممکن نیست.
تکنیک Pyrosequencing
پایه تکنیکهای توالی یابی نسل جدید، pyrosequencing است که ابتدائا به منظور بررسی SNPها مورد استفاده قرار میگرفت. به منظور استفاده در توالییابی، واکنشهای pyrosequencing به صورت پیدرپی (iterative pyrosequencing) انجام میگیرند تا توالی DNA را همزمان با سنتز آن ثبت نمایند. Pyrosequencing نیازی به پروسههای مربوط به جدا کردن قطعات مانند الکتروفورز ندارد و درنتیجه سریعتر انجام میشود.
در این تکنیک تعداد بازهایی که در هر آزمایش واحد توالی یابی میشود، حداکثر ۱۵۰ جفتباز است و این میزان در نگاه اول ممکن است پایین و درنتیجه ناکارآمد به نظر برسد؛ خصوصا اگر توالی یابی کل ژنوم مدنظرمان باشد. مزیتی که prosequencing دارد، امکان اجرای صدها تا هزاران واکنش به صورت موازی است. در نتیجه این کار، توالی بسیار سریعتر از توالی یابی خاتمه زنجیره سازی تعیین میگردد. به همین علت است که این تکنیک در حال جایگزینی روشهای پیشین در مطالعات ژنومی است.
Pyrosequencing نیز همانند تکنیکهای قبلی نیاز به تهیه DNA های الگوی تکرشتهای و یکسان دارد. پس از اتصال این مولکولها به پرایمر، توالی الگو توسط DNA پلیمراز و بدون افزودن دی دئوکسی نوکلئوتیدها همانندسازی میشود. همزمان با پیشرفت فرایند همانندسازی، نوکلئوتیدهای متصل شده شناسایی میگردند و درنتیجه خواندن توالی همزمان با پیشرفت واکنش صورت میگیرد.
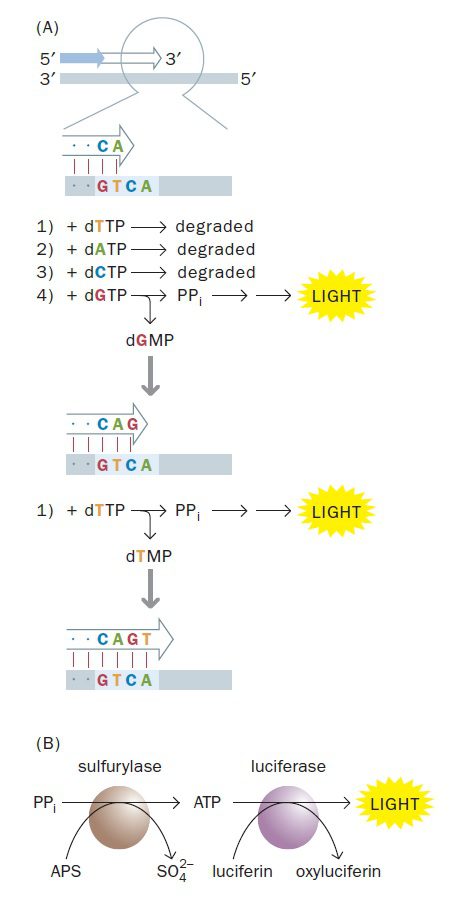
اساسیترین فرایند در pyrosequencing، شناسایی chemiluminescence است. همان طور که میدانید زنجیرههای DNA از مولکولهای dNTP ساخته میشوند و DNA پلیمراز باعث شکستن پیوند بین فسفات α و β میگردد. محصول نهایی dNMP حاوی فسفات α که به ساختار DNA الحاق میشود و نیز یک پیروفسفات متشکل از فسفات β و γ است. تکنیک pyrosequencing این ویژگی همانندسازی (آزاد شدن پیروفسفات) که در هر بار اضافه شدن نوکلئوتید به زنجیره رخ میدهد، را به کار میگیرد.
مجموعهای از واکنشها برای شناسایی پیروفسفات آزادشده به کار میروند. نخست، ATP سولفوریلاز پیروفسفات را در حضور آدنوزین ۵’ فسفوسولفات به ATP تبدیل میکند. سپس ATP تولیدشده در واکنشی شرکت میکند که در آن لوسیفراز، لوسیفرین را به اکسیلوسیفرین تبدیل میکند. اکسیلوسیفرین، ترکیبی است که متناسب با مقدار ATP آزاد شده نور مرئی ساطع مینماید. در نتیجه با هر بار الحاق نوکلئوتید به ساختار ژنوم، سیگنال نور شناسایی میگردد.
در تکنیک iterative pyrosequencing، dNTPها به صورت منفرد و پیدرپی عرضه میشوند. اگر هر چهار دئوکسی نوکلئوتید به یکباره اضافه میشدند، جرقههای نور همواره مشاهده میشد و هیچ داده مفیدی در مورد توالی مولکول موردنظر به دست نمیآمد. در صورتیکه dNTP ارائه شده، dNMP موردنیاز برای ادامه زنجیرهسازی را فراهم کند و پیروفسفات آزاد شود، نور تولید و توسط دوربین CCD ضبط میگردد. dNTP های مصرف نشده و ATP مازاد توسط آنزیم apyrase که در مخلوط واکنش وجود دارد و نوعی نوکلئوتیداز است، تجزیه میگردد. پس اگر dNTP ارائه شده در سنتز DNA استفاده نشود، هیچ نوری تولید نمیشود و apyrase، dNTP را تجزیه خواهد کرد. انجام این تکنیک به نظر پیچیده میرسد، اما چیزی که نیاز است فرایند ساده افزودن پیدرپی بازهاست که میتواند به سادگی ماشینی شود.
امروزه متدهای فراوانی به منظور Massively parallel sequencing ایجاد شدهاند که میتوانند میلیاردها واکنش توالی یابی را به طور همزمان پیش ببرند. نخستین تکنولوژیهایی توالی یابی نسل جدید از PCR به منظور تکثیر DNA هدف استفاده میکردند و به طور گسترده از سال ۲۰۰۷ موجود شدند. این تکنیکها در حال تغییر چهره علم ژنتیک مولکولی هستند و به طور گستردهای در توالی یابی مجدد (resequencing) سریع ژنومها به منظور اهداف مختلفی مانند توالی یابی ژنوم اشخاص و تعیین جهشها و نیز آنالیز ترانسکریپتوم مورد استفاده قرار میگیرند. ما در این قسمت دو مورد از آنها را که بیشتر از بقیه به کار میروند توضیح خواهیم داد: Illuminal و ۴۵۴ sequencing.
اساس هر دو متد تولید کتابخانههایی از قطعات DNA است که نشاندهنده کل ژنوم میباشند. به جای استفاده از سلولهای باکتریایی برای تولید این کتابخانهها، تکنیک PCR به منظور تولید میلیاردها عدد از این قطعات که هر یک به یک قطعه جامد متصل میشوند، به کار میرود. فرایند تکثیر به نحوی صورت میگیرد که نسخههای DNA حاصل از PCR به جای این که در محلول شناور باشند، در نزدیکی DNA اصلی به صورت متصل به سطح باقی میمانند. این فرایند تودههایی از قطعات DNA را تولید میکند که هر توده حاوی حدود هزار (سایر منابع؟؟؟؟) نسخه مشابه از بخش کوچکی از ژنوم است. این تودهها میتوانند در یک صفحه واحد جای گرفته و به صورت همزمان یا موازی (in parellel) توالی یابی شوند.

