تکنیک ligation-mediated PCR (LM-PCR) نوعی از PCR است که به منظور آنالیز ژنوم به کار رفته و علاوه بر مراحل عادی PCR، دارای مرحله اجباری DNA لیگاز نیز میباشد. تکنیک ligation-mediated PCR برای نخستین بار در footprinting ژنومی و واکنشهای توالییابی شیمیایی استفاده شد، ولی در موارد دیگری نیز قابل استفاده میباشد. طی این تکنیک، الیگونوکلئوتیدهای DNA کوچکی که linker و یا adaptor نامیده میشوند، به کار میروند که در ابتدا به قطعات DNA هدف متصل (ligated) میگردند. سپس پرایمرهای PCR که اختصاصی توالیهای linker هستند، به آنها متصل و برای تکثیر توالی هدف استفاده میشوند. این متد در توالییابی DNA و تعیین اولیه توالی نوکلئوتیدی DNA، تعیین الگوی متیلاسیون سیتوزین، تشکیل DNA lesion و ترمیم آن، DNA footpriniting و genome walking کاربرد دارد.
DNA ligase
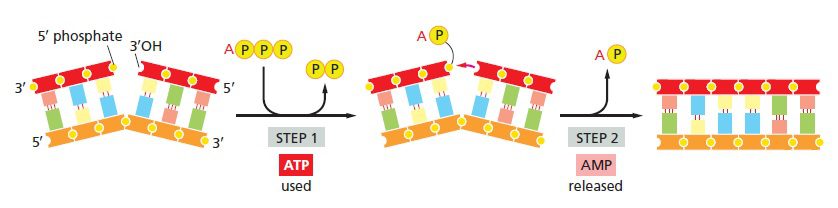
DNA مولکولی دورشتهای بوده و هر کدام از رشتههای آن دارای قطبیتی است که با انتهاهای ۳’ و ۵’ قسمتهای قندی مشخص میشود. دورشته جفتشده، غیرهمسو هستند و قطبیت آن ها در خلاف جهت یکدیگر قرار دارد. Nick که single strand break نیز خوانده میشود، به قسمتی از رشته DNA اطلاق میشود که دارای پیوند کوالانسی میان قند و باز مجاور آن نیست. Nickها میتوانند ناشی از آسیب DNA حین فرایند تکثیر نرمال DNA بوده و یا در اثر عمل آنزیم نوکلئاز ایجاد شوند. از آنجایی که وجود nickها پیوستگی رشته DNA را از میان میبرد، باید با تشکیل مجدد پیوند کووالانسی شکستهشده ترمیم شود تا دوباره مولکول DNA سالم حاصل آید. این بخشها خود سیگنالی را فراهم میکنند که سیستم proofreading را متوجه ترمیم رشته نیازمند آن میکند. ترمیم nickها با اتصال گروه ۳’-OH قند به فسفات مجاور آن که به گروه ۵’-OH قند بعدی اتصال دارد، انجام میگیرد و وظیفه آنزیم DNA لیگاز میباشد. DNA لیگاز با صرف انرژی، پیوند کووالانسی را مجددا برقرار کرده و تمامیت رشته را حفظ میکند. این موضوع از دو جنبه دارای اهمیت میباشد:
- Nickها در یک رشته وجود دارند و ترمیم آنها نیز در همان رشته انجام میگیرد؛ اما باید در نظر داشت که کل مولکول موردنظر دورشتهای است.
- بازهای رشته دارای nick، خصوصا آنهایی که بلافاصله nick را احاطه میکنند، باید به درستی با رشته مقابل جفت شده باشند. زیرا جایگاه فعال آنزیم لیگاز تنها در صورتی میتواند واکنش اتصال را کاتالیز کند که دو بخش موردنظر در اثر تشکیل جفتباز در کنار یکدیگر قرار داشته باشند.
اکنون تصور کنید که nick موردنظر به صورت طبیعی ایجاد نشده، و در عوض میان دو قطعه تکرشتهای سنتزشدهای از DNA قرار داشته باشد که مکمل ناحیه هدفی تکرشتهای هستند. اگر تستی داشته باشیم که طی آن برای تشکیل سیگنال باید دو رشته DNA به یکدیگر متصل شده و رشتهای بلندتر را تشکیل دهند، به این منظور میتوانیم از مرحلهای که به واسطه لیگاز انجام می شود استفاده کنیم. در این صورت است که میتوانیم وجود رشته مکمل این دو قطعه را بررسی نماییم. به عبارت دیگر، برای اینکه بدانیم آیا رشته A با توالی مشخص، در نمونه موجود است یا نه، میتوانیم دو رشته کوتاه، مجاور هم و مکمل A را که B و C نام دارند و nick آنها در مجاورت باز موردبررسی است، با نمونه مخلوط کنیم. این رشتهها به یکدیگر متصل شده و در صورتیکه انطباق کامل حاصل شود، سوبسترایی را برای عمل DNA لیگاز فراهم خواهند کرد. واکنش به وجود کوچکترین mismatch ها در نزدیکی nick، مانند SNPها، بسیار حساس بوده و مختل میشود. تنها در صورت انطباق کامل است که رشتههای B و C به یکدیگر پیوسته و رشته بلندتر B+C تولید میشود.
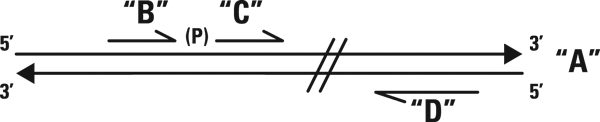
تشخیص ligation
اساس تکنیک LM-PCR اتصال الیگونوکلئوتید کوتاه شناختهشده به نام linker به انتهای ۵’ قطعات DNA موردنظر است که از آن انتها برش خوردهاند. برش میتواند در اثر آنزیم محدودکننده، به صورت شیمیایی (توالییابی شیمیایی)، تحت تاثیر UV و … ایجاد شود. Linker معمولا به توالی الیگودئوکسیریبونوکلوتیدی کوتاهی اطلاق میشود که معمولا دربردارنده جایگاه تشخیص آنزیم محدودکننده خاصی است. این توالی به انتهای مولکول DNA متصل شده و کلونینگ آن را تسهیل مینماید. به دنبال واکنش ligation، محصول توسط اندونوکلئاز تجزیه شده و قطعه DNA ای حاصل میشود که دارای انتهای کور یا چسبنده موردنظر است.
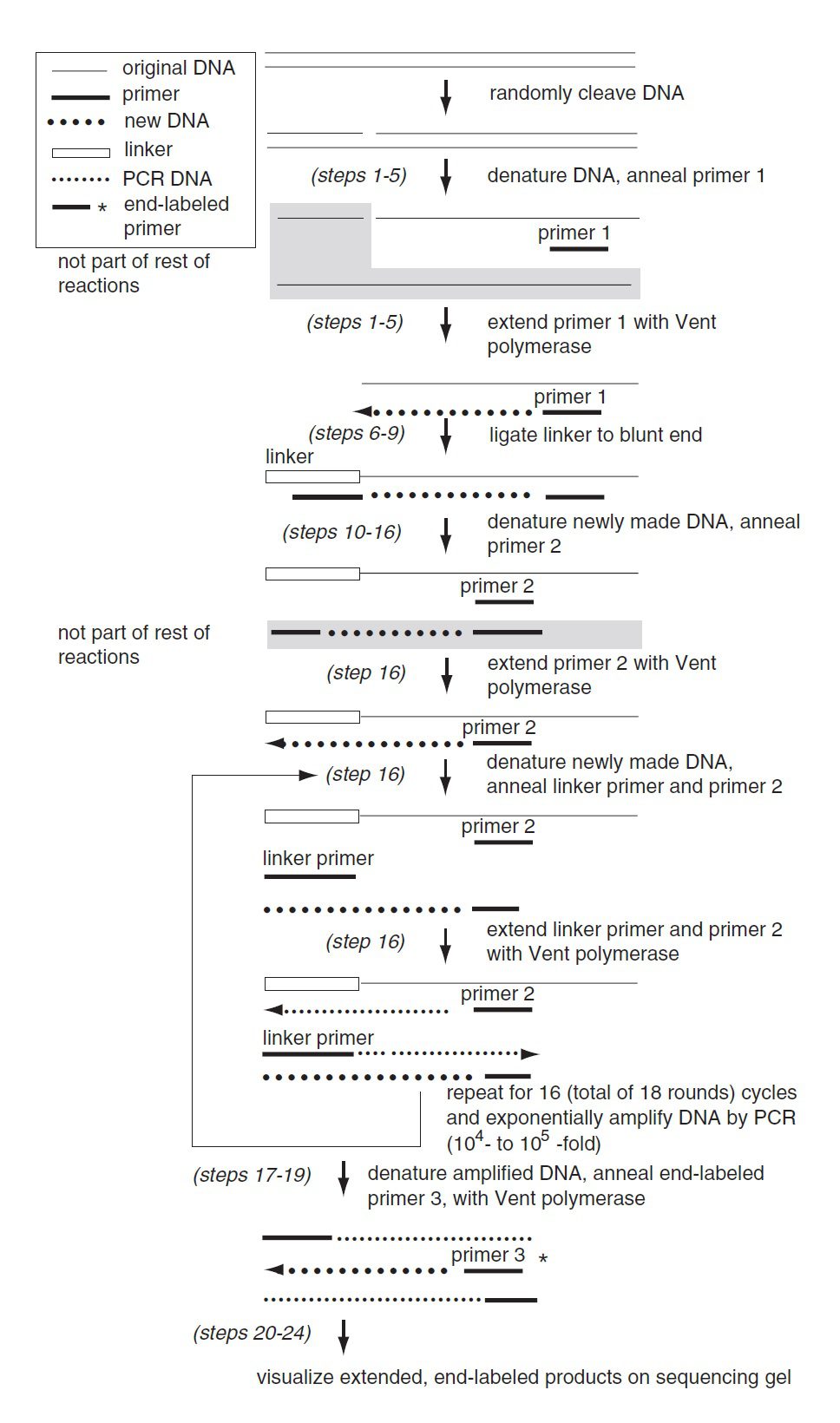
برای اینکه فاز نمایی واکنش PCR به انجام برسد، باید پرایمرها بتوانند ناحیه هدف را از هر دو سمت احاطه کنند. اتصال linker، توالی مشترکی را در همه انتهاهای ۵’ ایجاد کرده و امکان تولید سیگنال از طریق انجام فاز نمایی PCR را توسط linker به همراه پرایمر دیگری که اختصاصی ژن است و توالی آن را میدانیم، فراهم میکند. به عبارت دیگر، Ligation-mediated PCR تنها نیازمند دانستن توالی یک سمت است و پرایمر بعدی به توالی linker متصل خواهد شد. از آنجایی که در این حالت توالی مشخصی با طول معلوم به تمام قطعات متصل میگردد، میتوان جمعیتهای پیچیدهای از DNA مانند نردبانهای توالی را با حفظ رزلوشنی در حد یک جفتباز تکثیر نمود.
ماده آغازگر واکنش DNA ژنومی دارای nick است. این DNA به نحوی برش خورده که انتهای ۵’ فسفات خود را در محل برش حفظ کرده است. درنتیجه مجموعهای از قطعات را خواهیم داشت که از برش بخشی از DNA حین footprinting، واکنشهای توالییابی و … حاصل شدهاند. سپس این قطعات دناتوره شده و پرایمر اختصاصی ژن (پرایمر ۱) به ناحیه موردنظر اتصال مییابد. طی سنتز نخستین رشته، پرایمر توسط پلیمرازی پیشرونده (Vent DNA polymerase) تا محل برش گسترش داده میشود، تا اینکه قطعات DNA دوپلکس با انتهای کوردر یک سمت حاصل آیند.
در مرحله بعدی، DNA لیگاز اتصال linker تکجهتی (unidirectional) را به انتهای کور کاتالیز میکند. انتهای ۳’ رشته بلندتر ؟؟؟ linker به انتهای ۵’ قطعه DNA ژنومی اتصال مییابد (ligation). رشته کوتاهتر فاقد ۵’ فسفات بوده و درنتیجه به محصولات گسترشیافته توسط پرایمر اختصاصی ژن اتصال نمییابد. در مرحله سوم، DNA دناتوره شده، پرایمر اخصاصی ژن دوم (پرایمر ۲) به DNA ژنومی متصل میشود و توسط Vent DNA polymerase گسترش مییابد، تا زمانی که به linker برسد. میتوان از پرایمر ۱ نیز در این مرحله استفاده کرد. اما استفاده از پرایمر دوم background را کاهش خواهد داد.؟؟؟؟ پس پرایمرهای مورد استفاده در این مرحله عبارتند از: الیگونوکلئوتید بلندتر linker-primer و پرایمر دوم اختصاصی ژن.محصول گسترشیافته سوبسترایی ایدهآل برای PCR خواهد بود.
در یک سمت رشته حاصل، توالی linker قرار دارد و پرایمر linker میتواند به آن متصل شود. در سمت دیگر نیز توالی ژنومی متصلشونده به پرایمر اختصاصی ژن قرار دارد. تنها مولکولهایی که هر دو توالی را دارا هستند، حین واکنش PCR به صورت نمایی تکثیر خواهند شد. مولکولهایی که تنها یکی از توالیهای گفتهشده را دارند، به صورت خطی تکثیر میشوند. در ادامه پس از انجام ۲۰-۱۸ چرخه PCR، مرحله اختیاری تیمار با اگزونوکلئاز انجام میشود. هدف از فرایند گفتهشده، جدا کردن پرایمرها از واکنشهای پیشین است. در مرحله چهارم که مرحله گسترش نهایی است، پرایمر اختصاصی ژن سوم (پرایمر ۳) که با دومی دارای همپوشانی توالی است، به منظور لیبل کردن DNA به صورت غیرمستقیم به کار میرود. محصولات این مرحله که دارای انتهای لیبلشده هستند، روی ژل الکتروفورز مشاهده خواهند شد.
سوالی که ممکن است پیش بیاید، این است که چرا آنزیم لیگاز موجود در لوله واکنش به صورت رندوم محصولات موجود را به یکدیگر متصل نمیکند؟ علت آن است که آنزیمهای لیگاز مورداستفاده حساس به حرارت هستند و در طی دمای بالای نخستین دناتوراسیون PCR عملکرد خود را به طور دائمی از دست میدهند. خوشبختانه اکثر لیگازها مقاوم به حرارت نبوده و بسیاری از واکنشهای ligation دارای دمای اپتیمومی زیر دمای تمام مراحل PCR میباشند. درنتیجه نیازی به استفاده از هیچکدام از متدهای مهندسی آنزیم نخواهد بود.
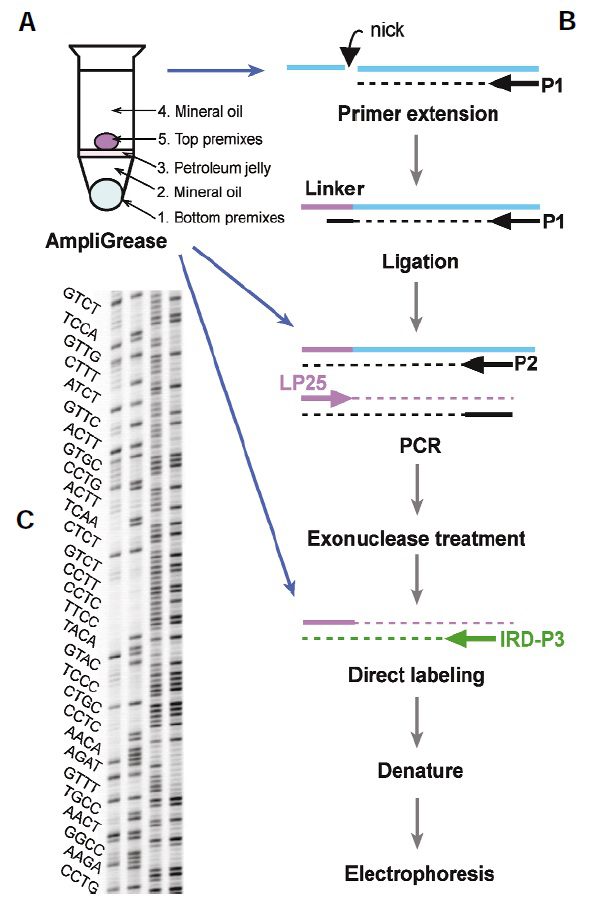
(B) LM-PCR (ref. 24) was automated by diluting products from each stage into the next utilizing AmpliGrease in three stages as indicated by the blue arrows. (C) Example of sequence of human TP53 exon-5 along upper stand. Lanes (from left to right) are G, G+A, C+T, and C products of Maxam–Gilbert25 sequencing reactions performed using HeLa cell DNA. The sequence displayed
lies 137–۲۵۱ bp from the primer sequence (P53R7028)