بسیاری از متدهای امروزی دستکاری DNA، RNA و پروتئینها، وابسته به وجود دانش قبلی راجع به توالی نوکلئوتیدی ژنوم موردنظر هستند. اما خود این توالیها برای نخستین بار چگونه شناخته شدهاند؟ و امروزه توالی مولکولهای DNA و RNA چگونه تعیین میگردد؟ تکنیک توالی یابی (sequencing) فرایند تعیین ترتیب دقیق بازها در نوکلئیکاسیدها است و باعث جهش ناگهانی در دانش ما از ژنتیک شده است. روشهای انجام هر چه سریعتر و آسانتر این فرایند عامل پیشبرنده پیشرفتهای اخیر بیوتکنولوژی است.
ایجاد کتابخانههای DNA پایه تکنیکهای توالی یابی ژنوم بود و انجام آن را امکان پذیر ساخت. در اواخر دهه ۱۹۷۰ بود که محققان تکنیکهایی را برای تعیین سریع و آسان توالیهای نوکلئوتیدی قطعات DNA خالص معرفی کردند. متدی که تاکنون بیشتر از همه مورد استفاده قرار گرفته است، تکینک توالی یابی Sanger نام دارد و اساس آن آنزیمی میباشد. طی این متد DNA پلیمراز با استفاده از توالیهای الگوی کلون شده و تکرشتهای، رشته جدید را سنتز میکند. توالی یابی Sanger زیربنای تمام تکنولوژيهای توالی یابی است که امروزه مورد استفاده قرار میگیرند و ژنومیکس (علم مطالعه ژنوم) را شکل داده است.
تکنیک Sanger به منظور تعیین توالی ژنومهای فراوانی مانند E. coli، مگس سرکه، کرمهای نواری (نماتودها)، موش و انسان مورد استفاده قرار گرفته است. Sanger و همکارانش نخستین دانشمندانی بودند که کل ژنوم یک جاندار را با استفاده از این تکنیک توالی یابی کردند. این ژنوم طولی در حدود ۵۰۰۰ باز داشت و متعلق به ویروسی به نام phiX174 بود. پس از این دستاورد آنان به تعیین توالی ژنومهای دیگری مانند ژنوم میتوکندری انسان ادامه دادند.
توالی یابی DNA با استفاده از تکنیک Sanger در توالی یابی ابتدایی ژنوم انسان بسیار کمک کننده بود. با این حال متدهای ابتدایی آهسته بودند، نیاز به نیروی انسانی فراوانی داشتند، هزینه تهیه مواد اولیه بالا بود، پیشرفت واکنشها نیاز به شرایط پیچیدهای داشت و بازده واکنشها نیز پایین بود. در نتیجه سالها طول میکشید که یک یا دو ژن توالی یابی شوند.
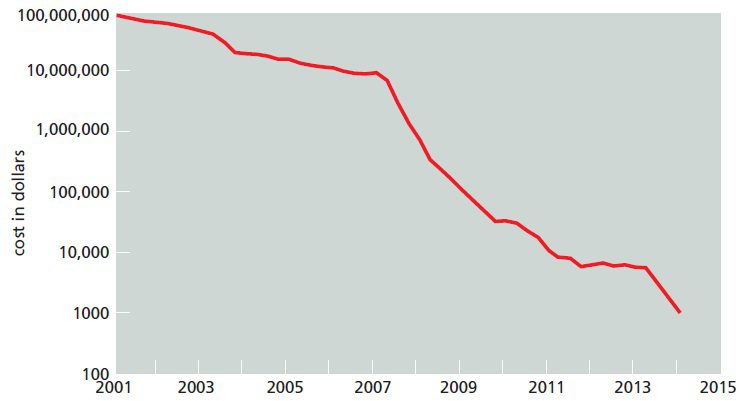
توالی مرجع ژنوم انسانی برای نخستین بار در سال ۲۰۰۳، با صرف یک میلیارد دلار هزینه و در طول ۱۳ سال تکمیل شد. در طول این پروژه هزینه تعیین توالی هر باز به علت وقوع پیشرفت در متد خاتمه زنجیر به همراه الکتروفورز موئین کاهش پیدا کرد و باعث شد که به بودجهای کمتر از حد انتظار نیاز داشته باشد. هزینه توالی یابی برای یک میلیون باز DNA در سپتامبر ۲۰۰۱، ۵۲۹۲ دلار و برای کل ژنوم انسان ۹۵ میلیون دلار بود.
در طی دهه اخیر، تکنولوژیهای توالی یابی پیشرفت فراوانی کردهاند؛ به نحوی که میتوان در عرض چند ساعت و با صرف هزینه کمتر، کل ژنوم انسان را که دارای طولی در حدود ۳.۲ بیلیون باز است، توالی یابی کرد. به طوری که تا اکتبر ۲۰۱۳ هزینه توالی یابی یک میلیون جفت باز به کمتر از ۶ سنت و درنتیجه هزینه توالی یابی کل ژنوم به حدود ۵۰۹۶ دلار رسید.
علت وقوع این کاهش چشمگیر، تکنیکهای توالی یابی نسل جدید (Next generation sequencing) بودند. معرفی توالی یابی نسل جدید در سال ۲۰۰۷ هیجانی در علم ژنومیکس پدید آورد و روشی کاملا جدید برای توالی یابی معرفی نمود. علاوه بر کاهش زمان و هزینه موردنیاز، تعداد ژنومهای تعیین توالی شده نیز با ابداع این تکنیکها افزایش قابل توجهی پیدا کرد. از جمله این متدها میتوان به تکنیک توالی یابی ۴۵۴ و Illumina اشاره کرد.
امروزه حتی متدهای جدیدتر، دقیقتر، ارزانتر و سریعتری نیز در حال توسعه هستند و تکنولوژیهای نسل سوم خوانده میشوند. این تکنیکها فاقد تمام مراحل تکثیر DNA هستند و توالی یک مولکول واحد DNA را تعیین میکنند. یکی از جدیدترین تکنیکهای توالی یابی نسل سوم، SMRT نام دارد و از جمله ویژگیهای خارقالعاده آن، کاهش نیاز به مواد اولیه گران قیمت، حساسیت بالا و پدید آوردن امکان مشاهده DNA پلیمراز هنگام ساخت رشته جدید است. استفاده از این تکنیک برای توالی یابی ژنوم انسان هنوز امکانپذیر نیست، اما اگر ممکن شود، خواهیم توانست به کمک آن کل ژنوم را در حدود یک ساعت توالی یابی کنیم. به احتمال زیاد در آینده شاهد توسعه متدهایی که حتی سریعتر و ارزانتر از متدهای کنونی هستند نیز خواهیم بود.
- تکنیک توالی یابی Sanger
- تکنیک توالی یابی نسل جدید
- تکنیک توالی یابی ۴۵۴
- تکنیک توالی یابی Illumina
- تکنیک توالی یابی نسل سوم
تکنیک توالی یابی Sanger
در سال ۱۹۷۴ Fredrick Sanger تکنیکی را به منظور تعیین توالی ژنها در محیط in vitro ابداع کرد. او به توالی آمینواسیدی انسولین علاقهمند بود و تصمیم گرفت که با استفاده از توالی نوکلئوتیدی آن به توالی آمینواسیدیاش برسد. متدی که Sanger و همکارانش در Medical Research Council Laboratory کمبریج اختراع کردند، روش خاتمه زنجیره سازی (Chain termination) نام گرفت و امروزه نیز کاربرد دارد. از جمله علل محبوبیت این تکنیک اتوماسیون آسان آن است.
توالی یابی Sanger همانند فرایند تکثیر DNA نیاز به پرایمر (رشته الیگونوکلئوتیدی کوتاهی از جنس RNA؟؟ که باید به نقطه یکسانی در تمام مولکولها متصل گردد)، DNA پلیمراز، توالی الگوی تکرشتهای و دئوکسی نوکلئوتیدها دارد. در طی واکنش، این مواد در محیط in vitro با یکدیگر مخلوط میشوند و پلیمراز نسخههای فراوانی را از توالی اصلی تولید میکند. اساس این تکنیک فرایند طبیعی همانندسازی DNA است و واکنش در حالت عادی میتواند تا پلیمریزه شدن هزاران نوکلئوتید ادامه پیدا کند؛ با این تفاوت که در این مورد همانندسازی در نقاطی قطع می شود و رشتههایی در اندازههای متفاوت به دست میآیند.
سوبسترای واکنش غالبا DNA نوترکیبی بود که در اثر دناتوراسیون توانایی اتصال به پرایمر توالییابی اختصاصی رشته را پیدا میکرد. قطعات DNA در وکتورهای فاژمیدی که در اثر دستکاری میتوانستند DNAهای نوترکیب تکرشتهای تولید کنند، نیز کلون میشدند. روش جایگزینی که امروزه نیز کاربرد فراوانی دارد، تولید قطعات DNA الگو با استفاده از PCR و تبدیل مولکولهای حاصل به فرم تک رشتهای است. در این حالت پلیمراز مورد استفاده نباید دارای خاصیت proofreading باشد تا سرعت الحاق نوکلئوتیدها افزایش یابد. محصول نهایی در تمام موارد ذکرشده، نسخههای فراوانی از مولکول DNA تکرشتهای موردنظر است.
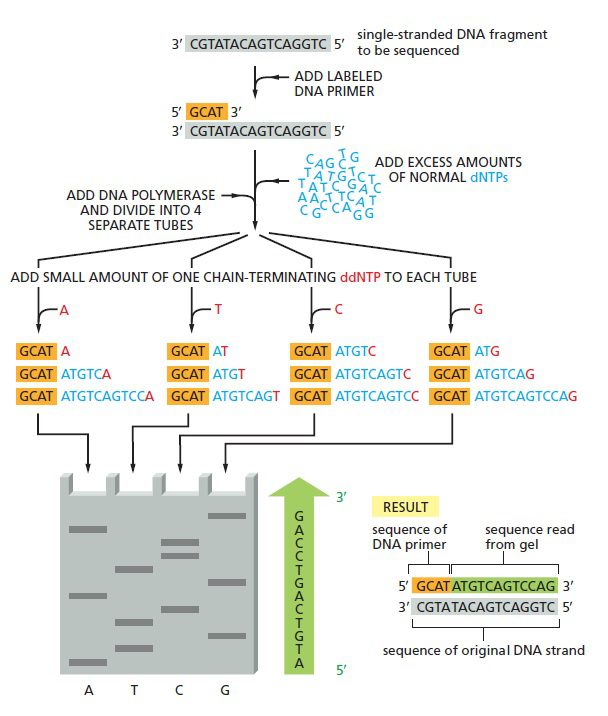
نخستین ترفند به دست آوردن توالی، متوقف کردن سنتز رشته جدید در هر کدام از جفتبازها است. این کار با مهار تصادفی سنتز زنجیره جدید DNA و درنتیجه تولید رشتههایی با طولهای متفاوت که میتوانند بر مبنای اندازه از یکدیگر جدا شوند، صورت میگیرد. در نتیجه تفاوت اندازه قطعات تولیدشده با یکدیگر در حدود یک جفت باز خواهد بود و در الکتروفورز نردبانی از قطعات به دست خواهد آمد. ترفند دوم تعیین ماهیت نوکلئوتید آخر هر قطعه است که اگر مشخص باشد، میتوان توالی را به طور مستقیم از روی ژل خواند. اما سوال این است که چگونه میتوان باز آخر هر قطعه را در نردبان توالی یابی تعیین کرد؟
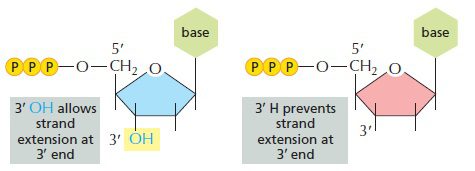
DNA پلیمراز سنتز رشته جدید DNA را بر اساس توالی رشته الگو انجام میدهد. زنجیره DNA حاوی دئوکسی نوکلئوتیدهایی است که دارای گروه هیدروکسیل در موقعیت ۳’ حلقه دئوکسی ریبوز میباشند. DNA پلیمراز افزودن نوکلئوتید بعدی را با اتصال فسفات روی کربن ۵’ آن، به ۳’-هیدروکسیل نوکلئوتید پیشین و با تشکیل پیوند فسفودیاستر انجام میدهد. در صورتیکه یکی از نوکلئوتیدها فاقد ۳’-هیدروکسیل باشد، نوکلئوتید دیگری افزوده نخواهد شد و زنجیره به طور ناگهانی خاتمه خواهد یافت.
در حین واکنش توالی یابی Sanger ، درصد معینی از نوکلئوتیدها که فاقد ۳’-هیدروکسیل هستند و دی دئوکسی نوکلئوتید (dideoxynucleotide: ddNTP) نامیده میشوند، با دئوکسی نوکلئوتیدهای نرمال مخلوط میشوند. آنزیمهای پلیمراز توانایی افتراق دئوکسی نوکلئوتیدها از دی دئوکسی نوکلئوتیدها را ندارند و این نوع از نوکلئوتیدها در صورت اتصال، به عنوان خاتمه دهنده زنجیره اختصاصی باز (base specific chain terminator) عمل خواهند کرد.
همان طور که گفته شد دئوکسی ریبونوکلئوتیدها در مقادیر بسیار بالاتری نسبت به دی دئوکسی ریبونوکلئوتیدها حضور دارند، در نتیجه خاتمه سنتز زنجیره جدید همواره در نزدیکی پرایمر رخ نمیدهد. در واقع ممکن است پلیمریزاسیون چند صد نوکلئوتید صورت بگیرد، پیش از آن که باز خاتمه دهنده رشته افزوده شود. در چنین واکنشهایی معمولا حداکثر طول قطعات ۸۰۰ نوکلئوتید خواهد بود.
تکنیک توالی یابی Sanger بر این پدیده استوار است که مولکولهای DNA تکرشتهای که تفاوتشان با یکدیگر تنها در حد یک نوکلئوتید است، میتوانند توسط الکتروفورز در ژل پلی آکریل آمید از یکدیگر جدا شوند. اساس این نوع از الکتروفورز با الکتروفورز در ژل آگارز یکسان است، با این تفاوت که حفرات در ژل پلی آکریل آمید کوچکتر هستند و در نتیجه امکان تفکیک قطعات کوچکتر را با دقت بالاتری پدید میآورند. در صورتی که این نوع از الکتروفورز از نوع Denaturing باشد، به علت وجود مقادیر بالای اوره و سایر ترکیبات دناتورهکننده مولکولهای DNA به صورت تکرشتهای باقی خواهند ماند.
نحوه خواندن توالی از پایین به بالا است؛ چون قطعاتی که نزدیک پرایمر خاتمه یافتهاندو سریعتر از مولکولهایی حرکت میکنند که در نقطهای دور از پرایمر جدا شده اند. نوارهای الکتروفورز به صورت نردبانی مشاهده میشوند که پلههای آن به اندازه یک نوکلئوتید از هم فاصله دارند. درنتیجه هر نوار نشانگر نوکلئوتیدی با توالی مکمل دی دئوکسی نوکلئوتیدی است که زنجیره را در آن نقطه خاتمه داده است. به عنوان مثال اگر دی دئوکسی نوکلئوتید A زنجیره را خاتمه داده باشد، نوکلئوتید موجود در رشته اصلی T است.
توالی یابی Sanger به صورت چهار واکنش موازی انجام میگیرد که هر کدام شامل ۴ نوع دئوکسی ریبونوکلئوتید (dATP، dCTP، dGTP، dTTP) به همراه درصد کمی از یکی از انواع دی دئوکسی نوکلئوتید میباشد. یکی از ۴ دئوکسی نوکلئوتید و یا پرایمر باید لیبل شده باشند تا رشته در حال سنتز نیزعلامتگذاری شود. با تنظیم غلظت دی دئوکسی نوکلئوتید در حدی بسیار پایینتر از آنالوگ دئوکسی نوکلئوتیدی آن، رقابتی بین این دو به منظور اتصال به رشته DNA در حال ساخت شکل خواهد گرفت. مقدار دئوکسی نوکلئوتید بسیار بیشتر است و با اتصال آن، سنتز DNA ادامه پیدا میکند، تا اینکه گاهی اوقات ناگهان با اتصال دی دئوکسی نوکلئوتید، پلیمریزاسیون متوقف و تولید زنجیره خاتمه مییابد.
از آن جایی که نمونه DNA شامل جمعیتی از مولکولهای یکسان است، هر یک از این چهار واکنش اختصاصی باز انتهای ۵’ مشترکی خواهند داشت که توسط پرایمر تعیین میشود. انتهای ۳’ در بین این قطعات متفاوت است، چون اتصال ddNTPها به صورت تصادفی و در یکی از نقاط فراوانی که قابلیت پذیرش آن باز را دارند انجام میشود.
اتوماسیون تکنیک توالی یابی Sanger کارایی آن را افزایش میدهد
در سالهای ابتدایی انجام این تکنیک، دانشمندان مجبور بودند خوانش توالیها را به صورت دستی و از روی اتورادیوگرام انجام دهند. هر کدام از انواع باز در ستونی جداگانه قرار داشت و خوانش اتورادیوگرام از پایین به بالا انجام میشد. دادهها نیز به صورت دستی وارد کامپیوتر میشدند. همان طور که میتوانید تصور کنید، این پروسه بسیار طولانی بود. ۱۲ ساعت برای الکتروفورز، ۱۲ ساعت برای ایجاد اتورادیوگرام و مدت زمان بسیار طولانیتری برای خواندن توالیها موردنیاز بود. علاوه بر آن احتمال وقوع اشتباه نیز بالا و استفاده از بازهای لیبل شده با مواد رادیواکتیو خطرناک بود.
با توجه به موارد گفته شده، انجام تعدادی از اصلاحات ضروری بودند، خصوصا اگر هدف توالی یابی ژنومهای بزرگتری مانند ژنوم انسان بود. ماشینهای توالی یابی اتوماتیک که از رنگهای فلورسنت استفاده میکردند، در اوایل دهه ۱۹۹۰ به صورت تجاری موجود شدند. انجام فرایند توالی یابی به صورت ماشینی جلوگیری از وقوع اشتباهات را آسانتر میکرد و سریعتر و امنتر نیز بود. با اینکه به علت گران قیمت بودن دستگاه آنالیزکننده توالی، هزینه راهاندازی این تکنیک بسیار بالا است، اما از آن جایی که چندین نمونه به صورت همزمان آنالیز میشوند، هزینه توالی یابی هر کدام از نمونهها بسیار پایین است.
در این فرایند، مخلوط واکنش شامل ۴ نوع دئوکسی نوکلئوتید، ۴ نوع دی دئوکسی نوکلئوتید، یک پرایمر، DNA الگو و DNA پلیمراز میباشد. به منظور تمایز دی دئوکسی نوکلئوتیدها از یکدیگر، از چهار رنگ فلورسنت متفاوت برای هر یک از چهار واکنش اختصاصی باز استفاده میشود. با انتخاب رنگهایی که دارای طول موج نشری متفاوتی هستند، میتوان هر چهار واکنش را به صورت یک نمونه در چاهک ژل وارد کرد.
تکثیر DNA الگو توسط PCR و در دستگاه Thermal cycler صورت میگیرد. ابتدا DNA الگو دناتوره میشود. سپس دما پایین آورده میشود تا پرایمرها متصل شوند. در نهایت دما تا حد مناسب برای فعالیت DNA پلیمراز افزایش مییابد تا قطعات موردنظر تکثیر یابند. حین پلیمریزاسیون، ممکن است دی دئوکسی نوکلئوتیدها متصل شده و زنجیره DNA خاتمه پیدا کند. نسبت دی دئوکسی نوکلئوتیدها به دئوکسی نوکلئوتیدها به گونهای تنظیم میشود که توقف همانندسازی حتما در هر کدام از بازهای A، G، T و C صورت بگیرد.
پس از آن که پلیمراز از توالی الگو هزاران نسخه که هر کدام در نوکلئوتید متفاوتی خاتمه یافتهاند، تهیه کرد، کل مخلوط در یک ستون الکتروفورز میشود. هنگام الکتروفورز، قطعات DNA از منبع تحریکی مانند لیزر عبور میکنند. در این حین، همزمان با عبور قطعات DNA از نقطهای معین در ژل، سیگنالهای فلورسنت شناسایی و ضبط میگردند. نتیجه، ایجاد یک intensity profile برای هر کدام از فلوروفورها است که در آن، هر کدام از چهار رنگ نشان دهنده باز متفاوتی است. همزمان اطلاعات به صورت الکترونیکی ذخیرهسازی میشوند.
تکنیکهای اولیه از ژل slab پلی آکریل آمید استفاده میکردند، اما با استفاده از توالی یابی capillary دانشمندان توانستند ظرفیت فرایند توالی یابی DNA را به میزان زیادی افزایش دهند. در این تکنیک نمونههای DNA از طریق لولههای شیشهای موئین بلند و بسیار نازکی که قطرشان در حدود ۰.۱ میلیمتر است و حاوی ژل پلی آکریل آمید هستند، الکتروفورز میشوند. انجام این کار باعث حصول به درجات بالاتری از اتوماسیون میگردد. برخی از ماشینهای توالی یابی میتوانند توالی بیش از ۳۸۴ نمونه مختلف DNA را با استفاده از لولههای موئین پر شده از ژل تشخیص دهند.
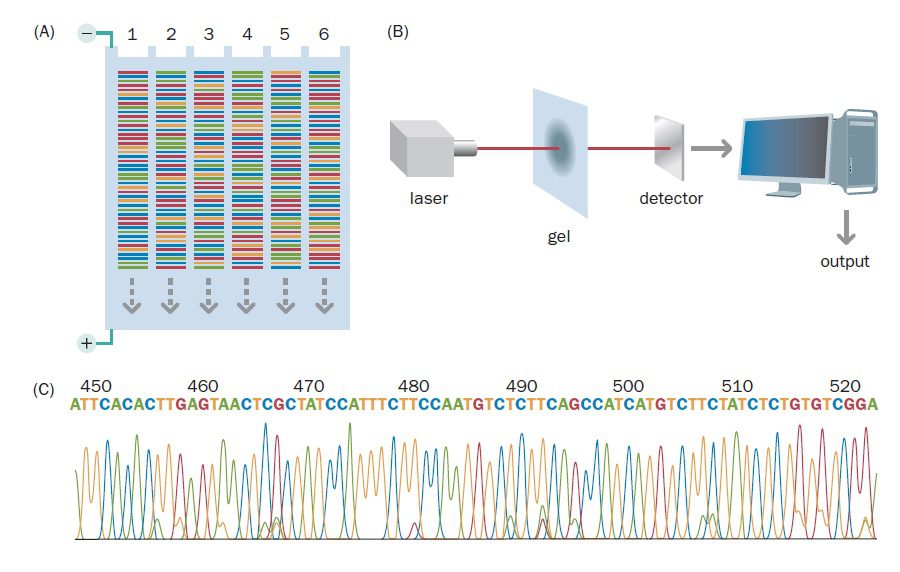
تکنیک توالی یابی Sanger با تغییرات جزئی که در آن ایجاد شد، به مدت سه دهه ژنتیک مولکولی را پایهریزی و ژنوم انسان و بسیاری از ارگانیسمهای دیگر را توالییابی نمود. با این حال نقصهایی نیز داشت و از جمله مهمترین آنها وابستگی آن به الکتروفورز برای جداسازی قطعات جدید DNA بود. این کار نه تنها نیاز تکنیک به نیروی انسانی را افزایش میدهد، بلکه توالی یابی تعداد زیادی از قطعات DNA را به صورت همزمان دشوار میکند. درنتیجه کارایی توالی یابی محدود و تقریبا ۳۰ تا ۶۰ کیلوباز در هر ۳ تا۴ ساعت انجام الکتروفورز است. این تکنیک همچنین سرعت پایین و هزینه بالایی دارد.
توالی یابی نسل جدید (NGS)
معرفی توالی یابی نسل جدید (Next Generation Sequencing= NGS) در سال ۲۰۰۷ تحولی در دنیای ژنومیکس پدید آورد، روشی کاملا جدید برای تعیین توالی DNA معرفی نمود و هزینه و زمان موردنیاز برای توالی یابی ژنوم را به شدت کاهش داد. مثالی که برای اهمیت این روش میتوان عنوان کرد آن است که پروژه ژنوم انسان ۱۰ سال طول کشید و بیش از ۳ میلیارد دلار بودجه مصرف کرد؛ این در حالی است که امروزه توالی یابی نسل جدید میتواند در عرض یک روز کل ژنوم انسان را با صرف هزینهای در حدود ۵۰۰۰ دلار توالی یابی کند. یکی از علل این موضوع آمادهسازی سریعتر نمونه نسبت به تکنیکهای پیشین است؛ به عنوان مثال دیگر نیازی به تولید کتابخانههای DNA در باکتریها نیست.علاوه بر صرفهجویی در هزینه و زمان، پس از کشف این تکنیکها تعداد ژنومهای توالی یابی شده نیز افزایش قابل توجهی داشته است. دو متد اصلی برای این نوع از توالی یابی وجود دارد: توالی یابی ۴۵۴ و Illumina.
این تکنیکها نیازی به الکتروفورز ندارند و ثبت توالی DNA را همزمان با سنتز آن از DNA الگوی تکرشتهای انجام میدهند. به عبارت دیگر در این تکنیکها، اتصال هر کدام از انواع نوکلئوتیدها به زنجیره DNA در حال رشد بررسی میشود. توالی یابیهای نسل جدید از پلتفرمهایی استفاده میکنند که میتوانند میلیونها قطعه DNA را در موقعیتهایی جداگانه ثابت و سپس آنالیز کنند و درنتیجه چندین ژنوم میتوانند به طور موازی در عرض کمتر از یک هفته توالی یابی شوند. همچنین این تکنیکها بسیار انعطافپذیر هستند و میتوانند برای تمام انواع و اندازههای مختلف ژنوم، از ویروس گرفته تا انسان به کار روند.
طبق موارد گفته شده، این تکنیکها این امکان را برای پژوهشگران پدید میآورد که ژنوم هزاران انسان را بررسی کنند، تفاوتهای موجود در توالی نوکلئوتیدی افراد در سراسر جهان را بیابند و پرده از جهشهایی که ریسک ابتلا به بیماریهای مختلف از سرطان تا اوتیسم را افزایش میدهند، بردارند. تکنیکهای توالی یابی نسل جدید همچنین امکان تعیین توالی ژنومی گونههای منقرض شده مانند نئاندرتالها و ماموتها را فراهم میکند. همچنین با توالییابی گونههای نزدیک به هم، درک اساس مولکولی رخدادهای کلیدی تکاملی در درخت زندگی، مانند چندسلولی شدن، بینایی و زبان آسانتر شده است. افزایش سرعت توالییابی تاثیراتی شگرف بر شاخههای مختلف بیولوژي و پزشکی گذاشته است؛ تا حدی که تصور وضعیت این علوم بدون آنها ممکن نیست.
تکنیک Pyrosequencing
پایه تکنیکهای توالی یابی نسل جدید، pyrosequencing است که ابتدائا به منظور بررسی SNPها مورد استفاده قرار میگرفت. به منظور استفاده در توالییابی، واکنشهای pyrosequencing به صورت پیدرپی (iterative pyrosequencing) انجام میگیرند تا توالی DNA را همزمان با سنتز آن ثبت نمایند. Pyrosequencing نیازی به پروسههای مربوط به جدا کردن قطعات مانند الکتروفورز ندارد و درنتیجه سریعتر انجام میشود.
در این تکنیک تعداد بازهایی که در هر آزمایش واحد توالی یابی میشود، حداکثر ۱۵۰ جفتباز است و این میزان در نگاه اول ممکن است پایین و درنتیجه ناکارآمد به نظر برسد؛ خصوصا اگر توالی یابی کل ژنوم مدنظرمان باشد. مزیتی که prosequencing دارد، امکان اجرای صدها تا هزاران واکنش به صورت موازی است. در نتیجه این کار، توالی بسیار سریعتر از توالی یابی خاتمه زنجیره سازی تعیین میگردد. به همین علت است که این تکنیک در حال جایگزینی روشهای پیشین در مطالعات ژنومی است.
Pyrosequencing نیز همانند تکنیکهای قبلی نیاز به تهیه DNA های الگوی تکرشتهای و یکسان دارد. پس از اتصال این مولکولها به پرایمر، توالی الگو توسط DNA پلیمراز و بدون افزودن دی دئوکسی نوکلئوتیدها همانندسازی میشود. همزمان با پیشرفت فرایند همانندسازی، نوکلئوتیدهای متصل شده شناسایی میگردند و درنتیجه خواندن توالی همزمان با پیشرفت واکنش صورت میگیرد.
اساسیترین فرایند در pyrosequencing، شناسایی chemiluminescence است. همان طور که میدانید زنجیرههای DNA از مولکولهای dNTP ساخته میشوند و DNA پلیمراز باعث شکستن پیوند بین فسفات α و β میگردد. محصول نهایی dNMP حاوی فسفات α که به ساختار DNA الحاق میشود و نیز یک پیروفسفات متشکل از فسفات β و γ است. تکنیک pyrosequencing این ویژگی همانندسازی (آزاد شدن پیروفسفات) که در هر بار اضافه شدن نوکلئوتید به زنجیره رخ میدهد، را به کار میگیرد.
مجموعهای از واکنشها برای شناسایی پیروفسفات آزادشده به کار میروند. نخست، ATP سولفوریلاز پیروفسفات را در حضور آدنوزین ۵’ فسفوسولفات به ATP تبدیل میکند. سپس ATP تولیدشده در واکنشی شرکت میکند که در آن لوسیفراز، لوسیفرین را به اکسیلوسیفرین تبدیل میکند. اکسیلوسیفرین، ترکیبی است که متناسب با مقدار ATP آزاد شده نور مرئی ساطع مینماید. در نتیجه با هر بار الحاق نوکلئوتید به ساختار ژنوم، سیگنال نور شناسایی میگردد.
در تکنیک iterative pyrosequencing، dNTPها به صورت منفرد و پیدرپی عرضه میشوند. اگر هر چهار دئوکسی نوکلئوتید به یکباره اضافه میشدند، جرقههای نور همواره مشاهده میشد و هیچ داده مفیدی در مورد توالی مولکول موردنظر به دست نمیآمد. در صورتیکه dNTP ارائه شده، dNMP موردنیاز برای ادامه زنجیرهسازی را فراهم کند و پیروفسفات آزاد شود، نور تولید و توسط دوربین CCD ضبط میگردد. dNTP های مصرف نشده و ATP مازاد توسط آنزیم apyrase که در مخلوط واکنش وجود دارد و نوعی نوکلئوتیداز است، تجزیه میگردد. پس اگر dNTP ارائه شده در سنتز DNA استفاده نشود، هیچ نوری تولید نمیشود و apyrase، dNTP را تجزیه خواهد کرد. انجام این تکنیک به نظر پیچیده میرسد، اما چیزی که نیاز است فرایند ساده افزودن پیدرپی بازهاست که میتواند به سادگی ماشینی شود.
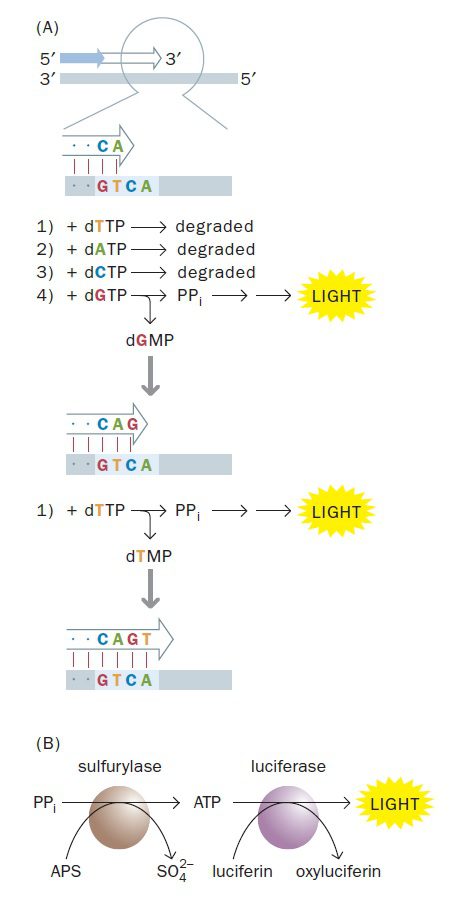
امروزه متدهای فراوانی به منظور Massively parallel sequencing ایجاد شدهاند که میتوانند میلیاردها واکنش توالی یابی را به طور همزمان پیش ببرند. نخستین تکنولوژیهایی توالی یابی نسل جدید از PCR به منظور تکثیر DNA هدف استفاده میکردند و به طور گسترده از سال ۲۰۰۷ موجود شدند. این تکنیکها در حال تغییر چهره علم ژنتیک مولکولی هستند و به طور گستردهای در توالی یابی مجدد (resequencing) سریع ژنومها به منظور اهداف مختلفی مانند توالی یابی ژنوم اشخاص و تعیین جهشها و نیز آنالیز ترانسکریپتوم مورد استفاده قرار میگیرند. ما در این قسمت دو مورد از آنها را که بیشتر از بقیه به کار میروند توضیح خواهیم داد: Illuminal و ۴۵۴ sequencing.
اساس هر دو متد تولید کتابخانههایی از قطعات DNA است که نشاندهنده کل ژنوم میباشند. به جای استفاده از سلولهای باکتریایی برای تولید این کتابخانهها، تکنیک PCR به منظور تولید میلیاردها عدد از این قطعات که هر یک به یک قطعه جامد متصل میشوند، به کار میرود. فرایند تکثیر به نحوی صورت میگیرد که نسخههای DNA حاصل از PCR به جای این که در محلول شناور باشند، در نزدیکی DNA اصلی به صورت متصل به سطح باقی میمانند. این فرایند تودههایی از قطعات DNA را تولید میکند که هر توده حاوی حدود هزار (سایر منابع؟؟؟؟) نسخه مشابه از بخش کوچکی از ژنوم است. این تودهها میتوانند در یک صفحه واحد جای گرفته و به صورت همزمان یا موازی (in parellel) توالی یابی شوند.

تکنیک توالی یابی ۴۵۴
Massively parallel pyrosequencing در سال ۲۰۰۵ توسط شرکت ۴۵۴ Life Sciences توسعه یافت و توسط Roche تجاریسازی شد. این تکنیک، توالی یابی ۴۵۴ نام دارد و نخستین تکنیک از توالی یابیهای نسل جدید است. توالی یابی ۴۵۴ میتواند بیش از یک میلیارد باز DNA را در یک روز تعیین توالی کند (معادل یک سوم ژنوم انسان). اولین گام در این تکنولوژي، آمادهسازی DNA به منظور تکثیر توسط PCR است. در واقع این تکنیکها به جای این که از محصولات PCR یا توالیهای کلونشده استفاده کنند، کار خود را با DNA ژنومی آغاز مینمایند. DNA ژنومی طبق پروتکل استاندارد استخراج DNA، از ارگانیسم موردنظر استخراج و سپس DNA خالص با استفاده از هموژنایزر اولتراسونیک (طی فرایند sonication) (و یا با استفاده از آنزیمهای محدودکننده)، به قطعات کوچکتری با اندازه ۳۰۰ تا ۵۰۰ جفتباز تقسیم میشود.
به منظور تکثیر هر کدام از قطعات، انتهایشان باید دارای توالی شناختهشدهای باشد و این موضوع خصوصا در ژنومهایی که هرگز توالی یابی نشدهاند، غیرممکن است. حتی اگر توالی شناخته شده باشد نیز sonication به صورت تصادفی DNA را میشکند و راهی برای شناخت دقیق توالی هر کدام از انتهاها وجود ندارد. راه حلی که برای دور زدن این مشکل به کار گرفته میشود، استفاده از linker ها یا adaptor ها است که قطعات DNA کوتاهی با توالی شناخته شده میباشند. اتصال دو adaptor متفاوت به انتهاهای قطعات DNA صورت میگیرد و پس از دناتوراسیون، مولکولهای تکرشتهای دارای adaptorهای متفاوت در دو انتها، گزینش میشوند. Adaptor ها دو وظیفه بر عهده دارند: نخست اینکه باعث اتصال قطعات به bead ها میشوند و دوم اینکه به عنوان مکانی برای اتصال پرایمر عمل میکنند. درنتیجه پرایمر یکسانی برای تمام قطعات میتواند استفاده شود.
مرحله بعدی، اتصال مولکولهای انتخاب شده به bead ها است و با استفاده از مکانیسم استرپتاویدین-بیوتین انجام میپذیرد. پروتئین باکتریایی استرپتاویدین تمایل اتصال بالایی به ویتامین بیوتین دارد و با طراحی یکی از دو adaptor به نحوی که در انتهای ۵’ خود دارای تگ بیوتین باشد، مولکولهای DNA به bead های پوشیده شده با استرپتاویدین متصل خواهند شد. به این نحو مولکولهای الگوی تکرشتهای DNA روی beadها ثابت میشوند و بعدا قطعات تولیدشده طی PCR نیز به سطح آن متصل خواهند شد.
جداسازی bead ها از یکدیگر با تشکیل امولسیون روغن در آب انجام میگیرد، طوری که هر قطره حاوی یک bead و واکنشدهندههای لازم برای PCR (دئوکسی نوکلئوتیدهای آزاد، پرایمرهای مکمل adaptor و Taq پلیمراز) باشد. این موضوع در این تکنیک بسیار حیاتی است. این قطرهها microreactor نام دارند. وجود امولسیون، مانع از پراکنده شدن مولکولهای DNA و واکنشدهندهها از یک bead به سایر beadها میگردد.
پس از اتمام PCR، حدود ده میلیون نسخه از هر قطعه DNA به صورت ثابتشده بر روی هر bead وجود خواهد داشت. در مرحله بعدی امولسیون از هم پاشیده میشود و beadها در چاههای picoliter که روی یک اسلاید قرار دارند، رسوب داده میشوند، به نحوی که هر چاه حاوی یک bead باشد. سطح تحتانی چاهها شفاف است و نور تولید شده میتواند از آن عبور کرده و توسط دستگاه شناسایی شود.
چاهها سپس با bead های کوتاهتری که سطحشان دارای ATP سولفوریلاز و لوسیفراز است، پوشانده میشوند. پیشسازهای dNTP یکبهیک و با ترتیب مشخصی (T، سپس A، سپس C، سپس G) به bead ها افزوده میشوند و همزمان با تکثیر، خوانش توالیها صورت میگیرد (sequencing by synthesis)؛ به این نحو که با هر بار اتصال نوکلئوتید درست، نور نشرشده در تمام واکنشها شناسایی و ضبط میگردد. شدت نور تولیدشده منطبق بر تعداد نوکلئوتیدهایی از یک نوع است که به رشته الگو متصل شدهاند. به عنوان مثال شدت نوری که در اثر اتصال سه باز A ایجاد میشود، سه برابر حالتی است که یک باز A وجود دارد.
تکنیک Massively parallel sequencing شرکت ۴۵۴ Life Sciences، در هر واکنش توالی یابی، توالیهای نسبتا بلندی را میخواند و به علاوه هزاران عدد از چنین واکنشهایی را به طور موازی انجام میدهد. در نتیجه محصول نهایی حدود ۱۰هزار بار بزرگتر از توالی یابی دی دئوکسی میگردد. البته ایراداتی نیز در این تکنیک وجود دارد؛ از جمله اینکه تشخیص تعداد بازها در حالتی که چندین باز تکراری به دنبال هم میآیند (مثلا AAAAAA) دشوار است.
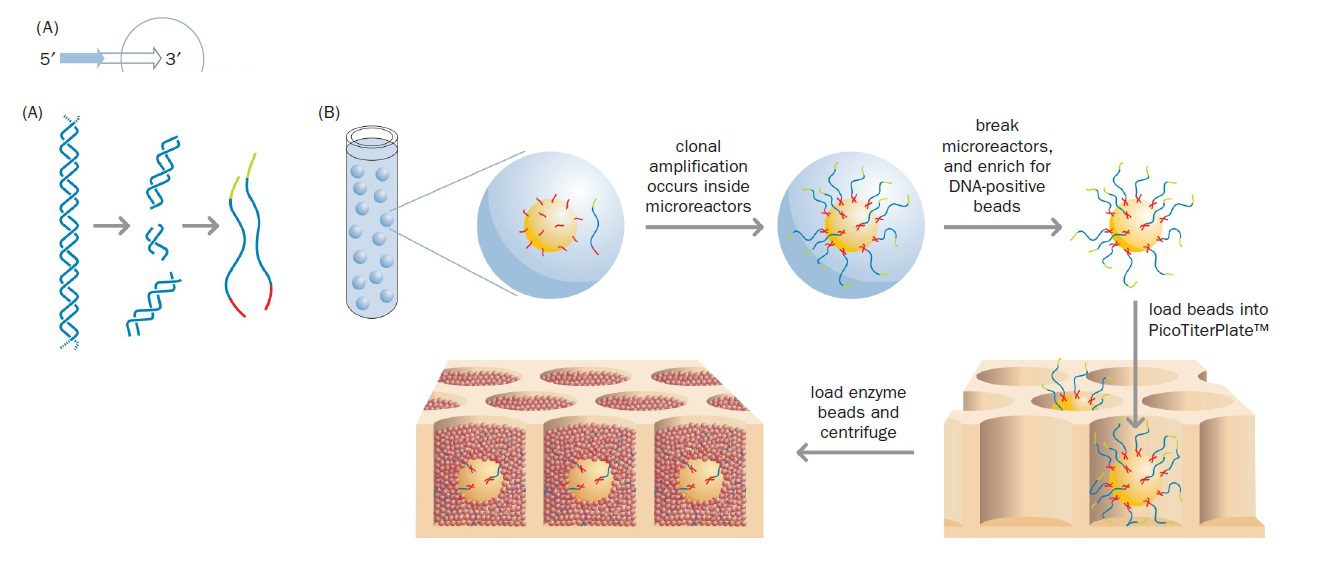
تکنیک توالی یابی Illumina
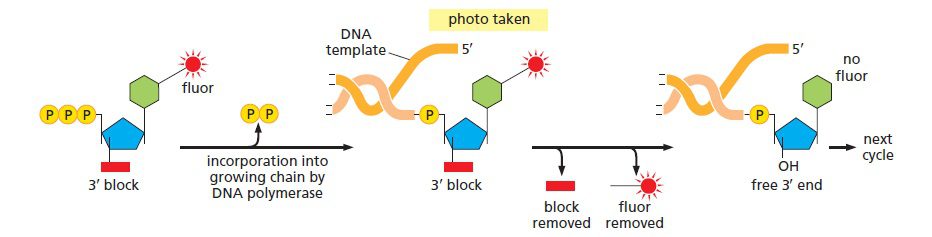
اساس تکنیک توالی یابی Illumina تکنیک دی دئوکسی است، اما از نوآوریهای فراوان دیگری نیز بهره برده است. در این تکنیک علیرغم پایین بودن تعداد توالیهای خوانده شده، محصولات بیشتری نسبت به تکنیک توالی یابی ۴۵۴ تولید میشود. طی این فرایند، از نوکلئوتیدهای متصل به یک مولکول فلورسنت قابل جداسازی که هر کدام از بازها رنگ متفاوتی دارند و نیز یک ماده شیمیایی خاتمه دهنده زنجیره استفاده میشود. به جای نبود گروه ۳’-OH که در توالی یابی در دئوکسی عادی باعث خاتمه زنجیره میشود، در این تکنیک نوکلئوتیدها دارای یک گروه شیمیایی هستند که میتواند ۳’-OH را بلاک کرده و از ادامه همانندسازی توسط DNA پلیمراز جلوگیری کند. این گروهها را میتوان به طریق آنزیمی جداسازی کرد.
همانند سایر تکنولوژيهای توالی یابی نسل جدید، اولین گام در این تکنولوژي، آمادهسازی DNA به منظور تکثیر توسط PCR است. در واقع این تکنیکها به جای این که از محصولات PCR یا توالیهای کلونشده استفاده کنند، کار خود را با DNA ژنومی آغاز مینمایند. DNA استخراج شده به قطعات کوچکتری که طولشان در حدود ۲۰۰ تا ۶۰۰ جفتباز است، شکسته میشود. توالیهای کوتاهی که adaptor نام دارند، به قطعات DNA متصل میگردند. سپس مولکولهای DNA دناتوره میشوند تا تکرشتهای شوند.
پس از طی این مراحل آمادهسازی، قطعات DNA به سطح یک فلوسل (flow cell) اتصال مییابند و سپس سطح فلوسل شسته میشود تا DNAهای متصل نشده در سطح باقی نمانند. سطح فلوسل دارای پرایمرهایی است که مکمل adaptorهای متصل شده به سطح میباشند. adaptorها و پرایمرها باید به حد کافی از هم دور باشند تا شناساگر موجود در کف فلوسل، آنها را در موقعیتهای جداگانهای تشخیص دهد.
مشابه توالی یابی ۴۵۴، قطعاتDNA ثابت شده در سطح فلوسل، در اثر انکوبه شدن با DNA پلیمراز و دئوکسی نوکلئوتیدها طی فرایندی به نام bridge sequencing همانندسازی میشوند. پرایمرهای موردنیاز برای تکثیر قطعات DNA به فلوسل متصل هستند؛ در نتیجه DNA در اثر اتصال به پرایمر، فرمی پل مانند پیدا میکند. این قطعات تکثیر و رهاسازی و سپس به منظور تکرشتهای شدن دناتوره میشوند تا تودهای از قطعات DNA مشابه را تشکیل دهند.
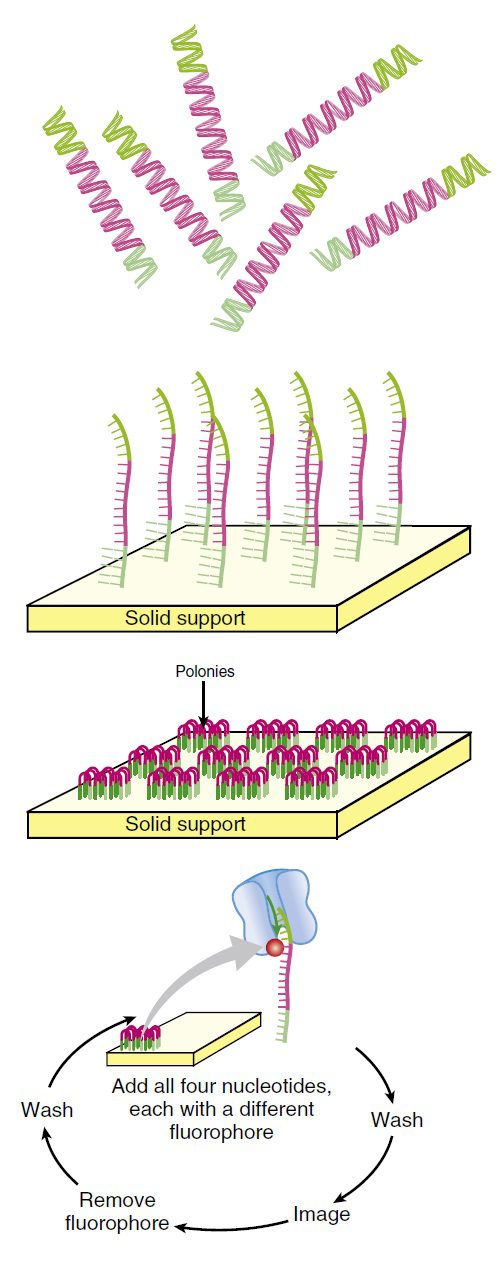
فرایند توالی یابی سپس به ترتیب زیر انجام میگیرد: چهار نوکلئوتید لیبل شده با رنگهای فلورسنت به همراه آنزیم DNA پلیمراز به میلیاردها توده DNA که روی یک اسلاید ثابت شدهاند، افزوده میشوند. در هر توده تنها نوکلئوتیدهای مکمل توالی الگو به صورت کوالانسی متصل و نوکلئوتیدهای متصل نشده شسته میشوند. سپس دوربینی دیجیتال با رزلوشن بالا، پس از تابیده شدن لیزر و فعال شدن رنگهای فلورسنت، تصویری را ثبت میکند که مشخص کننده نوکلئوتیدهای اضافه شده به زنجیره در هر توده است.
لیبل فلورسنت و گروه بلاککننده ۳’-OH به صورت آنزیمی جداسازی میشوند و فرایند به دفعات تکرار میگردد. به این نحو، میلیاردها واکنش توالی یابی به طور همزمان انجام میگیرند. با ردیابی تغییرات رنگ در هر توده، توالی DNA موجود در هر نقطه معین میشود. با اینکه هر کدام از توالیها به تنهایی نسبتا کوتاه هستند (حدود ۲۰۰ نوکلئوتید)، میلیاردها عدد از آنها به طور همزمان انجام میشوند تا در عرض یک روز، چندین ژنوم انسانی توالی یابی شود. بررسی باز به باز توالی باعث دقت بالای این متد شده است.
توالی یابی نسل سوم
موج جدیدی از تکنولوژیها که توالی یابی نسل سوم خوانده میشوند، و بسیار سریعتر از تکنیکهای پیشین هستند، در حال توسعه اند. برخی از این تکنیکها، تمام مراحل تکثیر DNA را دور میزنند و امکان تکثیر مولکولهای DNA واحدی که تکثیر نشدهاند را فراهم مینمایند. این کار از خطاهایی که هنگام تکثیر DNA رخ میدهند، جلوگیری میکند و یا آنها را به حداقل میرساند. همچنین تکنولوژیهای مورد استفاده در این تکنیکها بسیار سادهتر از متدهایی هستند که در فرایند آنها تکثیر توالی الگو وجود دارد و با صرف هزینه کمتری نیز انجام میشوند.
شرکت Helicos Biosciences نخستین تکنیک توالی یابی تک مولکولی، که به صورت تجاری موجود شد را ارائه کرد. دستگاه تولید شده توسط این کمپانی که HeliScopeTM نام داشت، در سال ۲۰۰۸ عرضه شد. از آنجایی که دستگاه توالی یاب HeliScope مولکولهای واحد را تشخیص میدهد، پس میتوان کل توالیهای مورد بررسی را روی سطح فلوسل به طور فشردهای جای داد (حدود ۱۰۰ میلیون الگوی تکمولکولی در هر سانتیمتر مربع و یا میلیاردها عدد در هر بار راهاندازی دستگاه). توالی یابی همزمان با واکنش سنتز صورت میگیرد و طی آن چندین چرخه افزودن تکنوکلئوتیدها به همراه مرحله شستوشو در بین چرخهها انجام میشود. در نتیجه میزان خوانش توالیها در هر بار ناچیز است، اما مجموع تمام توالیهای خوانده شده بسیار قابل توجه بوده و در حدود ۴۰ گیگاباز در هر بار راهاندازی به مدت ۸ روز میباشد. این میزان حتی ممکن است در آینده بیشتر نیز بشود. در این حالت، هزینه مواد اولیه نیز کمتر از توالی یابی قطعات تکثیرشده با PCR است.
تکنولوژیهای توالی یابی نسل سوم قدرتمندتری نیز در حال توسعه اند و به احتمال زیاد قرار است تحولی در فرایند توالی یابی DNA ، هم از لحاظ سرعت و هم از لحاظ هزینه، ایجاد کنند. تکنیک پیشرو در این مسیر، نوعی توالی یابی تک مولکولی DNA به نام SMRT (single-molecule real-time sequencing) است و توسط Pacific Biosciences توسعه یافته است. گفته میشود که این تکنولوژي خواهد توانست تعیین توالی را ۲۰۰۰۰ برابر سریعتر از تکنولوژیهای نسل جدید موجود در بازار انجام دهد.
تکنیک SMRT نیز توالی یابی را همزمان با سنتز DNA انجام میدهد، اما برخلاف متدهای پیشین، سنتز به صورت real-time انجام میگیرد و درنتیجه بسیار سریعتر است. SMRT میتواند در هر بار توالی یابی تکمولکول DNA، تعداد بازهای فراوانی را بخواند (۱۰ تا ۱۵ کیلوباز). تولید توالیهای بلند بسیار مهم است، چون در صورت بلندتر بودن قطعات، آسانتر میتوان توالی کل ژنوم را جمعآوری نمود. همچنین در مورد ژنومهای کوچک، کل توالی را به دست آورد، بی آن که مانند تکنولوژیهای قدیمیتر نیاز به متدهای وقتگیر و هزینهبر gap closing داشته باشیم.
به طور طبیعی در داخل سلولها، DNA پلیمرازها سنتز DNA جدید و دو برابر کردن ژنوم را در عرض چند دقیقه انجام میدهند. تکنیک SMRT به صورت real-time، آنزیمهای DNA پلیمرازی که سنتز مولکولهای DNA جدید از مولکول الگوی واحد که تکرشتهای است را انجام میدهند، تحت نظر میگیرد. در این حالت تشخیص نوکلئوتیدهای متصل شده به هر جایگاه با استفاده از نوکلئوتیدهای علامتگذاری شده با یکی از چهار فلوروفور متفاوت و طبق واکنش اختصاصیت باز انجام میشود.
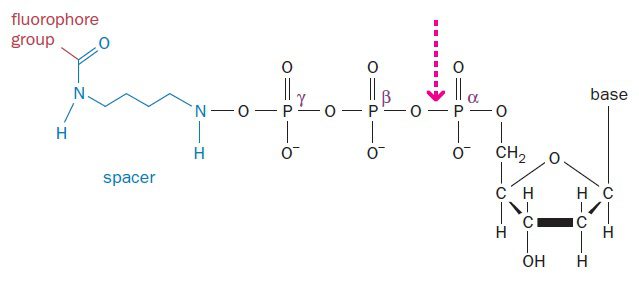
SMRT از دو نوآوری کلیدی بهره میگیرد. نخست اینکه فلوروفورها به طرز غیرمعمولی به dNTP ها لیبل شدهاند. در حالت عادی، فلوروفورها به نحوی به بازها متصل میشوند که با اتصال باز به ساختار DNA، جزئی دائمی از رشته DNA میگردند. در این شرایط، پس از افزوده شدن چندین نوکلئوتید به DNA، توده فلوروفورها به علت فضایی که اشغال میکند از سنتز DNA توسط آنزیم پلیمراز جلوگیری مینماید. به همین دلیل در بسیاری از متدهای توالی یابی که همزمان با سنتز صورت میگیرند، در هر بار، سنتز تنها یک نوکلئوتید انجام میشود و پس از آن واکنش خاتمه مییابد. در این حالت به مقادیر فراوانی از مواد اولیه نیاز خواهیم داشت و processivity آنزیم نیز بسیار محدود است.
در مقابل، در تکنیک SMRT، فلوروفورها به جای خود باز، به گروه فسفات γ که خارجیترین گروه در بخش تریفسفات نوکلئوتیدها است، متصل میشوند. هنگامی که چنین نوکلئوتیدی با باز مکملش در توالی الگو جفت میشود، میتوان سیگنال فلورسنت را پیش از برش گروه تریفسفات توسط پلیمراز، ضبط نمود؛ یعنی در لحظهای که dNTP هنوز متصل به جایگاه فعال آنزیم است. نوکلئوتیدهایی که مدت زمان طولانیتری به آنزیم متصل میمانند، مکمل هستند. سپس dNMP بدون لیبل، در اثر برش تولید شده و به ساختار ژنوم میپیوندد. در نتیجه پلیمراز میتواند بدون وقوع مشکلات مربوط به شکل فضایی به افزودن نوکلئوتیدها ادامه دهد و باعث تولید خوانشهای بلندی از توالی شود.
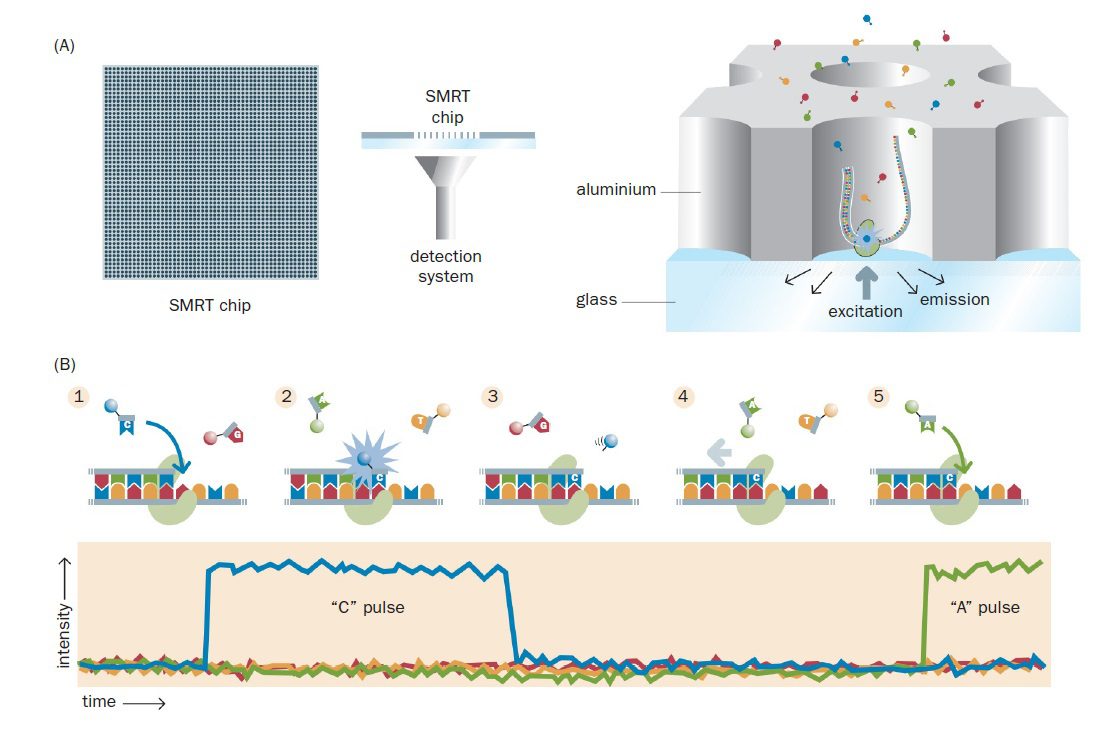
نوآوری مهم دوم، چیپ SMRT است و صفحه فلزی بسیار نازکی با پایه شیشهای میباشد. صفحه فلزی حدود ۱۰۰ نانومتری، حاوی یک میلیون حفره sub-wavelength به نام ZMW نانوفتونیک (nanophotonic zero-mode waveguide) است که قطری در حد یک دهم نانومتر دارند و هر کدام قادر به نگهداری یک مولکول DNA الگو میباشند. البته طبق توزیع Poisson، از یک میلیون حفره، تنها حدود ۳۳۰۰۰۰ عدد از آنها دارای توالی میگردند.
هر ZMW یک محفظه مصورسازی نانوفتونیک (nanophotonic visualization chamber) را تشکیل میدهد و DNA پلیمراز متصل به تکرشته الگو به کف پایه شیشهای متصل است. میتوان از روی پایه شیشهای ZMW، به طور مستقیم DNA پلیمراز را در حال انجام توالی یابی به صورت همزمان با همانندسازی، روی تکمولکول DNA مشاهده کرد. امواج نور جمعآوری شده از رشتههای در حال همانندسازی بررسی و به طور موازی آنالیز میشوند. با انجام یک سری اصلاحات، این تکنیک احتمالا خواهد توانست کل ژنوم انسان را در عرض یک ساعت و با صرف هزینهای در حدود صد دلار توالی یابی کند.
یکی دیگر از پیشرفتهای مهم در تکنیکهای توالی یابی نسل سوم ، توالی یابی nanopore است. موادی مانند سیلیکون میتوانند به نحوی شکل داده شوند که دارای حفرات بسیار ریزی گردند. در این حالت، DNA از چنین لوله باریکی عبور داده میشود (مشابه نخ کردن سوزن)؛ به نحوی که توالیهای مولکول تک رشتهای DNA به ترتیب از درون لوله عبور کنند. همزمان با این کار، مولکول DNA، بسته به ماهیت هر باز، تولید جریان الکتریکی متفاوتی میکند. الگوی جریانات میتواند برای استخراج توالی استفاده شود. اساس تولید جریان الکتریکی، مسدود شدن نانوپور در درجات و خصوصیات متفاوت توسط بازهای مختلف است.
با معرفی چنین تکنیکهای توالی یابی حساس و ارزانی، دانشمندان میتوانند شروع به توالی یابی مجدد ژنومها بکنند تا به دقت بالاتری دست یابند. به عنوان مثال، با استفاده از SMRT، توالی ژنوم E. coli با دقتی در حدود ۹۹.۹۹۹۹ درصد تعیین شده است. توالی یابی ژنوم انسان به این طریق هنوز ممکن نیست؛ اما زمانی که این امکان فراهم شود، احتمالا بتوانیم کل ژنوم را در حدود یک ساعت تعیین توالی کنیم و تمام این دستاوردها تنها پس از گذشت کمی بیش از یک دهه از اتمام پروژه ژنوم انسانی عملی گشته است!